Anatomie D’une polymérase – relation entre la fonction et la Structure
une réplication précise du génome est essentielle à la viabilité de tout organisme. Le concept général de copie de L’ADN était évident lors de l’élucidation de la structure double hélicoïdale de l’ADN et de l’identification de la complémentarité des paires de bases (1): un brin de nucléobases pourrait servir de modèle pour la synthèse d’un nouveau brin. Dans la décennie suivant ces découvertes, un agent a été purifié à partir d’E. coli qui a catalysé la duplication des brins D’ADN (2). Cet agent a été appelé une « polymérase ». E., l’ADN polymérase I de coli, la première ADN polymérase découverte n’était pas la polymérase réplicative primaire, mais plutôt une impliquée dans la résolution des fragments D’Okazaki et la réparation de l’ADN. Cela préfigurait les découvertes futures de nombreuses familles D’ADN polymérase, chacune répondant à des besoins cellulaires spécifiques pour la réplication et la réparation de l’ADN.
Les ADN polymérases servent d’enzymes fondamentales dans les sciences de la vie pour la même raison qu’elles sont de nature critique: elles copient L’ADN. Les applications de la polymérase comprennent l’étiquetage, le séquençage et l’amplification de l’ADN., Un protocole d’amplification spécifique, la réaction en chaîne par polymérase (PCR) est une technique largement utilisée qui utilise des polymérases thermophiles pour amplifier de manière exponentielle des segments D’ADN spécifiques (3) et permet une gamme d’applications allant du diagnostic humain et pathogène au clonage moléculaire dans les laboratoires de biologie du monde entier.
Polymérase Précision
PCR met les mêmes exigences de base et sur une polymérase comme une cellule met sur son système de réplication. Essentiellement, la polymérase doit être fiable, précise et rapide., La précision de la polymérase ou” fidélité » fait référence à la propension à incorporer le nucléotide correct tel que spécifié par le brin de gabarit. Les enzymes PCR standard sont, sans surprise, assez précises. Même L’ADN polymérase de Thermus aquaticus (Taq), considérée comme une polymérase PCR de faible fidélité, ne commet une erreur qu’une seule fois dans environ les insertions de nucléotides (12)., Les efforts de découverte et d’ingénierie de la polymérase ont produit des polymérases haute fidélité, qui commettent rarement des erreurs de substitution de base, nécessitant des méthodes de séquençage de l’ADN pour lire des millions de bases synthétisées afin de détecter toute erreur par la polymérase les progrès dans la mesure de la fidélité par séquençage à molécule unique ont identifié,
points de contrôle de Fidélité: sélection géométrique au site actif et au-delà
Les ADN polymérases assurent une réplication précise à l’aide d’une série de points de contrôle moléculaires, au site d’incorporation des nucléotides et au-delà (1). Lors de l’addition de nucléotides, le nucléotide entrant correct est positionné pour un alignement productif des groupes catalytiques, assurant une incorporation efficace. Cet alignement pour la catalyse est sensible aux distorsions de position causées par un appariement de base Watson-Crick incorrect, ce qui permet un décrochage cinétique à des paires de bases incorrectes ou non apparentées.,
relecture d’ADN polymérases
Une autre méthode d’augmentation de la fidélité consiste à ce que la polymérase ait une activité exonucléase 3→5, appelée « relecture”. En utilisant les points de contrôle moléculaires de base et de site actif décrits ci-dessus, L’ADN polymérase Taq est incroyablement précis, mais les enzymes de relecture peuvent avoir une fidélité encore plus élevée. Cette précision supplémentaire est transmise par relecture, la polymérase « vérifiant » si le nucléotide correct a été inséré dans le gabarit., Si une non-concordance est détectée, l’ADN est transféré du domaine de polymérisation à un domaine exonucléase N-terminal 3→5 de la polymérase. Le nucléotide mal incorporé est excisé, L’ADN retourne dans le domaine de polymérisation et la copie peut reprendre (Figure 2).
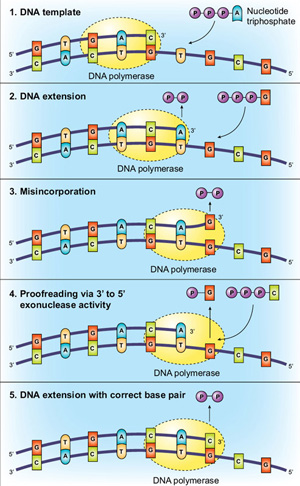
le bactériophage T4 s’est avéré être un système expérimental utile pour évaluer l’importance de l’activité exonucléase 3→5 pour une réplication précise de l’ADN (6). Des Mutations dans le gène T4 43 (qui Code l’ADN polymérase) ont été identifiées qui ont diminué ou augmenté la fidélité. En définissant un rapport d’activité exonucléase/polymérase (N/P) pour une enzyme mutante, on a constaté que les polymérases ayant de faibles rapports N/P étaient plus sujettes aux erreurs que celles ayant des rapports N/P élevés., Une explication de cette observation est que lors de l’incorporation d’une base non appariée, il est plus probable que l’exonucléase enlèvera le nucléotide avant que l’activité de la polymérase ne l’étend dans des enzymes ayant des rapports N/P plus élevés. Fait intéressant, l’efficacité de la relecture d’une polymérase peut montrer une dépendance à la séquence. Par exemple, les séquences AT-rich sont plus efficacement relues que les régions GC. On pense que cet écart est dû à la stabilité plus faible des régions AT qui facilite la fusion des brins et, par conséquent, l’activité de relecture.,
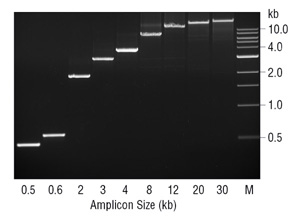
l’absence d’activité exonucléase 3→5 peut avoir des ramifications autres que la fidélité dans la PCR. L’absence d’activité de relecture dans L’ADN polymérase Taq a été proposée pour limiter la taille de l’amplicon possible avec cette enzyme (7). Généralement, Taq fonctionne mieux lors de l’amplification de fragments D’ADN < 2 KO, et peut fonctionner avec des fragments jusqu’à 3-4 Ko. Lorsqu’il est maintenu à cette taille d’amplicon, le Taq est une enzyme robuste et facilement optimisée. Cependant, au-dessus de ~3 Ko, son efficacité diminue rapidement., Au cours de la PCR, Taq va mal incorporer les nucléotides et produire des discordances, à laquelle il est sujet au décrochage et est plus susceptible de se dissocier avant de s’étendre par rapport aux 3 extrémités appariées correctement. Par conséquent, à une certaine taille d’amplicon et un taux d’erreur de polymérase suffisamment incompatibles 3 extrémités peuvent s’accumuler pour inhiber efficacement le processus de PCR. Ces 3 extrémités incompatibles sont particulièrement problématiques pour Taq car il manque l’activité exonucléase 3→5 pour les éliminer., En ajoutant une petite quantité d’enzyme de relecture telle que L’ADN polymérase Deep Vent®, l’amplification des fragments ≥ 20 kb peut être obtenue (Figure 3). Étant donné que la grande majorité de l’enzyme dans le mélange est Taq ADN polymérase, il fait probablement la majeure partie de l’extension de l’amorce, avec la relecture de la polymérase à évent profond éliminant les incompatibilités inhibitrices 3 générées par Taq.,
Processivité de la polymérase
l’importance de l’activité de relecture en PCR est largement connue depuis près de deux décennies, mais une autre propriété, la processivité, a attiré une attention accrue., « Processivité » est un terme qui fait référence au nombre de nucléotides incorporés par une polymérase en un seul événement de liaison (avant dissociation). Taq ADN polymérase ajoute environ 50 nucléotides par événement de liaison (8). Pourquoi est-ce important? Une polymérase à faible processivité ou « distributive” étend une population de modèles d’une manière sensiblement différente d’une polymérase processive. Une polymérase hautement distributive se lie à un modèle, ajoute quelques nucléotides et se dissocie, laissant une population de modèles qui peut être étendue également avec le temps., Une polymérase hautement processive lie un modèle et s’étend avec des événements de liaison plus longs.
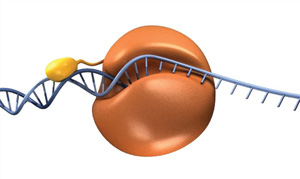
Il s’ensuivrait qu’avec suffisamment de temps, le résultat d’une réaction de polymérase processive ou distributive serait une population de modèles copiés. Cependant, dans certaines circonstances, il est possible que la polymérase processive ait des performances supérieures. La sous-unité α de la polymérase III D’E. coli, qui fait partie de la polymérase réplicative principale, présente une processivité de < 10 paires de bases et une vitesse de < 20 nucléotides/seconde (nt/s)., Cependant, lorsque la sous-unité s’associe aux autres sous-unités du réplisome, en particulier la pince coulissante, la processivité effective et la vitesse de réplication augmentent à > 50 Ko et 1 000 nt/s, respectivement (9). Le terme « processivité efficace » est utilisé parce qu’il existe des données indiquant que la sous-unité polymérase peut s’échanger dans le réplisome, mais que le réplisome maintient une réplication rapide et processive de l’ADN (10).
pour tirer parti de la processivité en PCR, les chercheurs ont fusionné un domaine de liaison de l’ADN à une polymérase archéale (11)., Cette enzyme chimérique possède plusieurs propriétés améliorées, mais elle est notamment capable d’amplifier l’ADN avec des temps d’extension plus courts et de produire plus efficacement des produits D’ADN plus longs, raccourcissant ainsi les temps de thermocyclage globaux. Cette idée de fusion est à la base de L’ADN polymérase Haute Fidélité Q5 et de L’ADN polymérase Haute Fidélité Phusion®, deux polymérases disponibles chez NEB (Figure 4).
les Orientations Futures
de Nombreuses propriétés affectent l’efficacité et l’utilité d’une PCR polymerase. L’architecture du site actif de la polymérase et l’activité de relecture affectent la précision du produit final. Les mélanges de polymérases et la fusion avec une protéine de liaison à L’ADN confèrent des performances PCR supérieures pour la longueur de l’amplicon et, dans le cas de la chimère, la vitesse de réaction., D’autres avancées importantes dans la PCR, telles que les polymérases à démarrage à chaud pour augmenter la spécificité de la réaction, la PCR multiplex (Figure 5) et le qPCR ont également révolutionné les sciences de la vie.
comme l’ont démontré les mélanges d’ingénierie et les chimères, les propriétés de la polymérase elle-même peuvent être modulées pour améliorer les performances de la PCR. À l’avenir, il est probable que les propriétés de la polymérase seront de plus en plus adaptées à des applications PCR spécifiques, et c’est donc un domaine de recherche important à L’ONÉ.,

Voir le tableau de sélection de L’ADN polymérase de L’ONÉ















