Anatomie einer Polymerase-Wie Funktion und Struktur zusammenhängen
Eine genaue Genomreplikation ist entscheidend für die Lebensfähigkeit eines Organismus. Das allgemeine Konzept für das Kopieren von DNA zeigte sich bei der Aufklärung der doppelhelikalen Struktur der DNA und der Identifizierung der Komplementarität von Basenpaaren (1): Ein Strang von Nukleobasen könnte als Vorlage für die Synthese eines neuen Strangs dienen. Innerhalb eines Jahrzehnts dieser Entdeckungen wurde ein Mittel aus E. coli gereinigt, das die DNA-Strangverdopplung katalysierte (2). Dieses Mittel wurde als „Polymerase“bezeichnet. E., coli-DNA-Polymerase I, die erste DNA-Polymerase, die entdeckt wurde, war nicht die primäre replikative Polymerase, sondern eine, die an der verzögerten Strang-Okazaki-Fragmentauflösung und DNA-Reparatur beteiligt war. Dies deutete auf zukünftige Entdeckungen vieler DNA-Polymerasefamilien hin, die jeweils spezifischen zellulären Anforderungen für die DNA-Replikation und-reparatur dienen.
DNA-Polymerasen dienen als grundlegende Enzyme in den Biowissenschaften aus dem gleichen Grund, aus dem sie kritisch sind: Sie kopieren DNA. Polymerase-Anwendungen umfassen DNA-Kennzeichnung, Sequenzierung und Amplifikation., Ein spezifisches Amplifikationsprotokoll, die Polymerase-Kettenreaktion (PCR), ist eine weit verbreitete Technik, die thermophile Polymerasen zur exponentiellen Amplifikation spezifischer DNA-Segmente (3) einsetzt und eine Reihe von Anwendungen von der Human-und Pathogendiagnostik bis zum molekularen Klonen in Biologielabors auf der ganzen Welt ermöglicht.
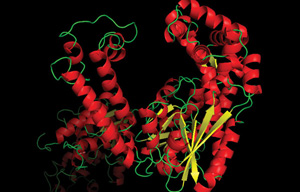
Genauigkeit der Polymerase
PCR stellt die gleichen grundlegenden Anforderungen an eine Polymerase wie eine Zelle an ihr Replikationssystem. Im Wesentlichen sollte die Polymerase zuverlässig, genau und schnell sein., Polymerase Genauigkeit oder“ Treue “ bezieht sich auf die Neigung, das richtige Nukleotid zu integrieren, wie durch den Schablonenstrang angegeben. Die standard-PCR-Enzyme sind, nicht überraschend, ziemlich genau. Selbst die DNA-Polymerase von Thermus aquaticus (Taq), die als PCR-Polymerase mit niedriger Wiedergabetreue gilt, macht bei ungefähr Nukleotideinfügungen nur einmal einen Fehler (12)., Polymerase-Entdeckung und Engineering-Bemühungen haben High-Fidelity-Polymerasen hergestellt, die selten Basensubstitutionsfehler machen, DNA-Sequenzierungsmethoden erfordern Millionen von synthetisierten Basen zu lesen, um Fehler durch die Polymerase Fortschritte bei der Messung der Treue durch Einzelmolekül-Sequenzierung zu erkennen identifiziert Q5® High-Fidelity-DNA-Polymerase als Fidelity 280X größer thanTaq DNA-Polymerase (12).,
Fidelity Checkpoints: Geometrische Selektion an der aktiven Stelle und darüber hinaus
DNA-Polymerasen gewährleisten eine genaue Replikation unter Verwendung einer Reihe molekularer Checkpoints an der Stelle des Nukleotideinbaus und darüber hinaus (1). Während der Nukleotidaddition wird das korrekt ankommende Nukleotid für eine produktive Ausrichtung der katalytischen Gruppen positioniert, um eine effiziente Einarbeitung zu gewährleisten. Diese Ausrichtung für die Katalyse ist empfindlich gegenüber Positionsverzerrungen, die durch eine falsche Watson-Crick-Basenpaarung verursacht werden, was ein kinetisches Abwürgen bei falschen oder nicht verwandten Basenpaaren ermöglicht.,
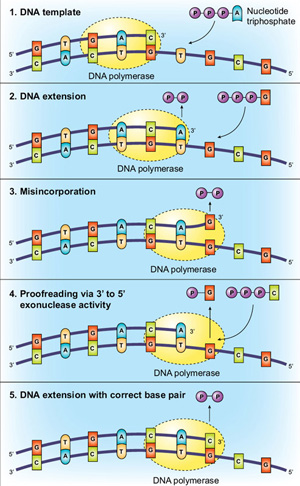
Korrekturlesen von DNA-Polymerasen
Eine weitere Methode zur Erhöhung der Treue besteht darin, dass die Polymerase eine 3→5-Exonuklease-Aktivität aufweist, die als „Korrekturlesen“bezeichnet wird. Unter Verwendung der oben beschriebenen molekularen Kontrollpunkte für Basenpaarung und aktive Stelle ist die Taq-DNA-Polymerase unglaublich genau, aber Korrekturlesen von Enzymen kann eine noch höhere Wiedergabetreue aufweisen. Diese zusätzliche Genauigkeit wird durch Korrekturlesen vermittelt, wobei die Polymerase „prüft“, ob das richtige Nukleotid in die Schablone eingefügt wurde., Wenn eine Nichtübereinstimmung festgestellt wird, wird die DNA von der Polymerisationsdomäne auf eine N-terminale 3→5-Exonukleasedomäne der Polymerase übertragen. Das falsch eingearbeitete Nukleotid wird ausgeschnitten, DNA bewegt sich zurück in die Polymerisationsdomäne und das Kopieren kann fortgesetzt werden (Abbildung 2).
Bacteriophage T4 erwies sich als nützliches experimentelles System zur Bewertung der Bedeutung der 3→5-Exonuklease-Aktivität für eine genaue DNA-Replikation (6). Mutationen im T4-Gen 43 (das die DNA-Polymerase kodiert) wurden identifiziert, die entweder die Treue verringerten oder erhöhten. Durch die Definition eines Exonuklease/Polymerase (N/P) – Aktivitätsverhältnisses für ein mutiertes Enzym wurde festgestellt, dass Polymerasen mit niedrigen N/P-Verhältnissen fehleranfälliger waren als solche mit hohen N / P-Verhältnissen., Eine Erklärung für diese Beobachtung ist, dass es beim Einbau einer nicht übereinstimmenden Base wahrscheinlicher ist, dass die Exonuklease das Nukleotid entfernt, bevor die Polymeraseaktivität es in Enzymen mit höheren N/P-Verhältnissen ausdehnt. Interessanterweise kann die Korrekturlesewirksamkeit einer Polymerase Sequenzabhängigkeit zeigen. Zum Beispiel werden AT-Rich-Sequenzen effektiver korrekturlesen als GC-Regionen. Es wird angenommen, dass diese Diskrepanz auf die geringere Stabilität von AT-Regionen zurückzuführen ist, die das Strangschmelzen und damit die Korrekturlesen erleichtert.,
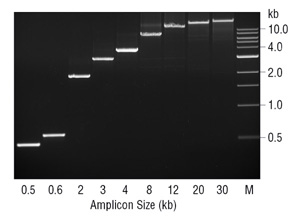
Das Fehlen einer Exonukleaseaktivität von 3→5 kann andere Verzweigungen als die in der PCR haben. Der Mangel an Korrekturleseaktivität in Taq-DNA-Polymerase wurde vorgeschlagen, um die mit diesem Enzym mögliche Amplicongröße zu begrenzen (7). Im Allgemeinen ist Taq bei der Amplifikation von DNA-Fragmenten < 2 kb am besten geeignet und kann mit Fragmenten bis zu 3-4 kb arbeiten. Wenn Taq auf diese Amplicongröße gehalten wird, ist es ein robustes, leicht optimiertes Enzym. Oberhalb von ~3 kb sinkt jedoch schnell die Wirksamkeit., Während der PCR wird Taq Nukleotide falsch integrieren und Fehlanpassungen erzeugen, bei denen es zum Abwürgen neigt und eher dissoziiert, bevor es sich im Vergleich zu den Basenpaar-3-Enden ausdehnt. Daher kann sich bei einer bestimmten Amplicongröße und Polymerasefehlerrate genug nicht übereinstimmende 3 Enden ansammeln, um den PCR-Prozess effektiv zu hemmen. Diese nicht übereinstimmenden 3 Enden sind besonders problematisch für Taq, weil es die 3→5 Exonuklease Aktivität fehlt, um sie zu entfernen., Durch Zugabe einer kleinen Menge korrekturleser Enzyme wie Deep Vent ® DNA Polymerase kann eine Amplifikation von Fragmenten ≥ 20 kb erreicht werden (Abbildung 3). Da die überwiegende Mehrheit des Enzyms in der Mischung Taq-DNA-Polymerase ist, macht es wahrscheinlich den Großteil der Primerverlängerung, wobei die Korrekturlesung Deep Vent Polymerase die von Taq erzeugten inhibitorischen 3-Fehlanpassungen entfernt.,
Polymerase Processivity
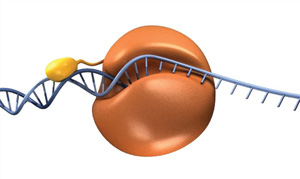
Die Bedeutung von Korrekturlesen für PCR ist seit fast zwei Jahrzehnten weithin bekannt, aber eine andere Eigenschaft, processivity, hat erhöhte Aufmerksamkeit erlangt., „Prozessivität“ ist ein Begriff, der sich auf die Anzahl der Nukleotide bezieht, die von einer Polymerase in ein einzelnes Bindungsereignis (vor der Dissoziation) eingebaut werden. Taq-DNA-Polymerase fügt ungefähr 50 Nukleotide pro Bindungsereignis hinzu (8). Warum ist das wichtig? Eine Low-Processivity-oder“ distributive “ Polymerase erweitert eine Population von Vorlagen auf eine merklich andere Weise als eine Processivpolymerase. Eine hochverteilende Polymerase bindet an eine Schablone, fügt ein paar Nukleotide hinzu und dissoziiert und hinterlässt eine Population von Schablonen, die mit der Zeit gleichmäßig erweitert werden können., Eine hochprozessive Polymerase bindet eine Vorlage und erweitert sie mit längeren Bindungsereignissen.
Es würde folgen, dass das Ergebnis einer prozessiven oder distributiven Polymerase-Reaktion bei ausreichender Zeit eine Population kopierter Vorlagen wäre. Unter bestimmten Umständen ist es jedoch möglich, dass die prozessive Polymerase eine überlegene Leistung aufweist. Die E. coli-Polymerase-III-α-Untereinheit, Teil der hauptreplikativen Polymerase, weist eine Prozessivität von < 10 Basenpaaren und eine Geschwindigkeit von < 20 Nukleotiden/Sekunde (nt/s) auf., Wenn die Untereinheit jedoch mit den anderen Replisomuntereinheiten, insbesondere der Gleitklemme, assoziiert wird, erhöhen sich die effektive Prozessivität und Replikationsgeschwindigkeit auf > 50 kb bzw. Der Begriff „effektive Prozessivität“ wird verwendet, weil Daten vorliegen, die darauf hindeuten, dass die Polymerase-Untereinheit im Replisom austauschen kann, das Replisom jedoch eine schnelle, prozessive DNA-Replikation beibehält (10).
Um die Prozessivität in der PCR auszunutzen, haben Forscher eine DNA-Bindungsdomäne mit einer archaealen Polymerase verschmolzen (11)., Dieses chimäre Enzym hat mehrere verbesserte Eigenschaften, aber insbesondere ist es in der Lage, DNA mit kürzeren Verlängerungszeiten zu amplifizieren und längere DNA-Produkte effizienter zu produzieren, wodurch die gesamten Thermozykluszeiten verkürzt werden. Diese Fusionsidee basiert auf Q5 High-Fidelity DNA Polymerase und Phusion® High-Fidelity DNA Polymerase, zwei von NEB erhältlichen Polymerasen (Abbildung 4).
Future Directions
Viele Eigenschaften beeinflussen die Wirksamkeit und den Nutzen einer PCR-Polymerase. Polymerase Active Site Architecture und Korrekturlesen beeinflussen die Genauigkeit des Endprodukts. Polymerasemischungen und Fusion zu einem DNA-Bindungsprotein verleihen eine überlegene PCR-Leistung für die Amplikonlänge und im Fall der Chimäre die Reaktionsgeschwindigkeit., Andere wichtige Fortschritte in der PCR, wie Hot-Start-Polymerasen zur Erhöhung der Reaktionsspezifität, Multiplex-PCR (Abbildung 5) und qPCR, haben auch die Biowissenschaften revolutioniert.
Wie durch technische Mischungen und Chimären demonstriert, können die Eigenschaften der Polymerase selbst moduliert werden, um die PCR-Leistung zu verbessern. In Zukunft werden die Polymerase-Eigenschaften wahrscheinlich zunehmend auf spezifische PCR-Anwendungen zugeschnitten sein, und als solches ist dies ein wichtiges Forschungsgebiet bei NEB.,

Zeigen Sie das DNA-Polymerase-Auswahldiagramm von NEB an















