Biology for Majors I
Lernziele
- Beschreiben Sie die Rolle der RNA-polymerase
in Diesem Abschnitt wird erweitert, nach der spezifischen Rolle von RNA-Polymerasen während der Transkription. Lesen Sie weiter, um die Rolle von RNA-Polymerasen in jeder Transkriptionsstufe zu lernen.,
Beginn der Transkription
Im Gegensatz zur prokaryotischen Polymerase, die selbst an eine DNA-Schablone binden kann, benötigen Eukaryoten mehrere andere Proteine, sogenannte Transkriptionsfaktoren, um zuerst an die Promotorregion zu binden und dann die entsprechende Polymerase zu rekrutieren.
Die drei eukaryotischen RNA-Polymerasen
Die Merkmale der eukaryotischen mRNA-Synthese sind deutlich komplexer als die von Prokaryoten. Anstelle einer einzelnen Polymerase, die fünf Untereinheiten umfasst, haben die Eukaryoten drei Polymerasen, die jeweils aus 10 Untereinheiten oder mehr bestehen., Jede eukaryotische Polymerase erfordert auch einen bestimmten Satz von Transkriptionsfaktoren, um sie in die DNA-Vorlage zu bringen.
Die RNA-Polymerase I befindet sich im Nucleolus, einer spezialisierten nuklearen Unterstruktur, in der ribosomale RNA (rRNA) transkribiert, verarbeitet und zu Ribosomen zusammengesetzt wird (Tabelle 1). Die rRNA-Moleküle werden als strukturelle RNAs betrachtet, da sie eine zelluläre Rolle spielen, aber nicht in Protein übersetzt werden. Die rRNAs sind Bestandteile des Ribosoms und für den Translationsprozess essentiell. RNA-Polymerase I synthetisiert alle rRNAs mit Ausnahme des 5S-rRNA-Moleküls., Die Bezeichnung “ S „gilt für“ Svedberg “ – Einheiten, einen nichtditiven Wert, der die Geschwindigkeit charakterisiert, mit der sich ein Teilchen während der Zentrifugation sediert.
| Tabelle 1.,f Transcription | α-Amanitin Sensitivity | |||
|---|---|---|---|---|
| I | Nucleolus | All rRNAs except 5S rRNA | Insensitive | |
| II | Nucleus | All protein-coding nuclear pre-mRNAs | Extremely sensitive | |
| III | Nucleus | 5S rRNA, tRNAs, and small nuclear RNAs | Moderately sensitive | |
RNA polymerase II is located in the nucleus and synthesizes all protein-coding nuclear pre-mRNAs., Eukaryotische Prä-mRNAs werden nach der Transkription, jedoch vor der Übersetzung einer umfangreichen Verarbeitung unterzogen (Abbildung 1). Zur Verdeutlichung wird in der Diskussion dieses Moduls über Transkription und Übersetzung in Eukaryoten der Begriff „mRNAs“ verwendet, um nur die reifen, verarbeiteten Moleküle zu beschreiben, die zur Übersetzung bereit sind. RNA-Polymerase II ist für die Transkription der überwiegenden Mehrheit der eukaryotischen Gene verantwortlich.

Abbildung 1. Eukaryotische mRNA enthält Introns, die herausgespleißt werden müssen. Eine 5 ‚Kappe und 3‘ Poly-A Schwanz werden ebenfalls hinzugefügt.,
Die RNA-Polymerase III befindet sich ebenfalls im Kern. Diese Polymerase transkribiert eine Vielzahl von strukturellen RNAs, die die 5S-Prä-rRNA, Transfer-Prä-RNAs (Prä-tRNAs) und kleine nukleare Prä-RNAs umfassen. Die tRNAs spielen eine entscheidende Rolle bei der Übersetzung; Sie dienen als Adaptermoleküle zwischen der mRNA-Schablone und der wachsenden Polypeptidkette. Kleine nukleare RNAs haben eine Vielzahl von Funktionen, einschließlich des „Spleißens“ von Prä-mRNAs und der Regulierung von Transkriptionsfaktoren.,
Ein Wissenschaftler, der ein neues Gen charakterisiert, kann bestimmen, welche Polymerase es transkribiert, indem er testet, ob das Gen in Gegenwart eines bestimmten Pilzgiftes α-Amanitin exprimiert wird (Tabelle 1). Interessanterweise beeinflusst α-Amanitin, das von Amanita Phalloiden, dem Todeskappenpilz, produziert wird, die drei Polymerasen sehr unterschiedlich. Die RNA-Polymerase I ist gegenüber α-Amanitin völlig unempfindlich, was bedeutet, dass die Polymerase DNA in vitro in Gegenwart dieses Giftes transkribieren kann. Im Gegensatz dazu ist RNA-Polymerase II extrem empfindlich gegenüber α-Amanitin und RNA-Polymerase III ist mäßig empfindlich., Die Kenntnis der transkribierenden Polymerase kann einen Forscher auf die allgemeine Funktion des untersuchten Gens hinweisen. Da die RNA-Polymerase II die überwiegende Mehrheit der Gene transkribiert, werden wir uns in unseren nachfolgenden Diskussionen über eukaryotische Transkriptionsfaktoren und Promotoren auf diese Polymerase konzentrieren.
RNA-Polymerase-II-Promotoren und Transkriptionsfaktoren
Eukaryotische Promotoren sind viel größer und komplizierter als prokaryotische Promotoren. Beide haben jedoch eine Sequenz ähnlich der vorherigen Sequenz von Prokaryoten., In Eukaryoten wird diese Sequenz TATA Box genannt und hat die Konsenssequenz TATAAA auf dem kodierenden Strang. Es befindet sich bei -25 bis -35 Basen relativ zur Initiationsstelle (+1) (Abbildung 2). Diese Sequenz ist nicht identisch mit der E. coli-Filterbox, behält jedoch das A–T-reiche Element bei. Die Thermostabilität von A-T-Bindungen ist gering und dies hilft der DNA-Schablone, sich in Vorbereitung auf die Transkription lokal abzuwickeln.,
Anstelle des einfachen σ-Faktors, der die prokaryotische RNA-Polymerase an ihren Promotor bindet, stellen Eukaryoten einen Komplex von Transkriptionsfaktoren zusammen, die erforderlich sind, um RNA-Polymerase II zu einem proteinkodierenden Gen zu rekrutieren. Transkriptionsfaktoren, die an den Promotor binden, werden Basaltranskriptionsfaktoren genannt. Diese basalen Faktoren werden alle als TFII (für Transkriptionsfaktor/Polymerase II) plus einen zusätzlichen Buchstaben (A-J) bezeichnet. Der Kernkomplex ist TFIID, das ein TATA-bindendes Protein (TBP) enthält., Die anderen Transkriptionsfaktoren fallen systematisch auf die DNA-Schablone, wobei jeder den Pre-Initiation-Komplex weiter stabilisiert und zur Rekrutierung von RNA-Polymerase II beiträgt.
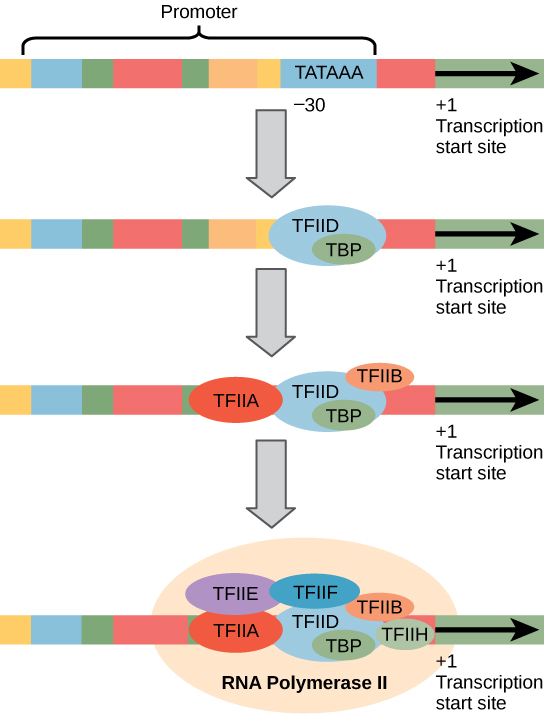
Abbildung 2. Ein generalisierter Promotor eines durch RNA-Polymerase II transkribierten Gens ist gezeigt. Transkriptionsfaktoren erkennen den Promotor. RNA-Polymerase II bindet dann und bildet den Transkriptionsinitiationskomplex.,
Praxisfrage
Ein Wissenschaftler spleißt einen eukaryotischen Promotor vor ein bakterielles Gen und fügt das Gen in ein bakterielles Chromosom ein. Würden Sie erwarten, dass die Bakterien das Gen transkribieren?
Einige eukaryotische Promotoren haben auch eine konservierte CAAT-box (GGCCAATCT) bei etwa -80. Weiter vor der TATA-Box können eukaryotische Promotoren auch eine oder mehrere GC-reiche Boxen (GGCG) oder Octamer-Boxen (ATTTGCAT) enthalten., Diese Elemente binden zelluläre Faktoren, die die Effizienz der Transkriptionsinitiation erhöhen, und werden häufig in „aktiveren“ Genen identifiziert, die ständig von der Zelle exprimiert werden.
Basaltranskriptionsfaktoren sind entscheidend für die Bildung eines Präinitiationskomplexes auf der DNA-Schablone, der anschließend RNA-Polymerase II zur Transkriptionsinitiierung rekrutiert. Die Komplexität der eukaryotischen Transkription endet nicht mit den Polymerasen und Promotoren., Eine Armee anderer Transkriptionsfaktoren, die an Upstream-Enhancer und Schalldämpfer binden, hilft auch dabei, die Häufigkeit zu regulieren, mit der Prä-mRNA aus einem Gen synthetisiert wird. Enhancer und Schalldämpfer beeinflussen die Effizienz der Transkription, sind jedoch nicht erforderlich, damit die Transkription fortgesetzt werden kann.
Die Evolution von Promotoren
Die Evolution von Genen kann ein bekanntes Konzept sein. Mutationen können in Genen während der DNA-Replikation auftreten, und das Ergebnis kann für die Zelle von Vorteil sein oder auch nicht., Durch die Veränderung eines Enzyms, Strukturproteins oder eines anderen Faktors kann der Mutationsprozess Funktionen oder physikalische Merkmale transformieren. Eukaryotische Promotoren und andere genregulatorische Sequenzen können sich jedoch ebenfalls entwickeln. Betrachten Sie zum Beispiel ein Gen, das über viele Generationen für die Zelle wertvoller wird. Vielleicht kodiert das Gen ein Strukturprotein, das die Zelle für eine bestimmte Funktion im Überfluss synthetisieren muss. Wenn dies der Fall ist, wäre es für die Zelle von Vorteil, dass der Promotor dieses Gens Transkriptionsfaktoren effizienter rekrutiert und die Genexpression erhöht.,
Wissenschaftler, die die Entwicklung von Promotorsequenzen untersuchen, haben unterschiedliche Ergebnisse gemeldet. Dies liegt zum Teil daran, dass es schwierig ist, genau abzuleiten, wo ein eukaryotischer Promotor beginnt und endet. Einige Promotoren treten innerhalb von Genen auf; Andere befinden sich sehr weit stromaufwärts oder sogar stromabwärts der Gene, die sie regulieren. Als die Forscher ihre Untersuchung jedoch auf menschliche Kernpromotorsequenzen beschränkten, die experimentell als Sequenzen definiert wurden, die den Präinitiationskomplex binden, stellten sie fest, dass sich Promotoren noch schneller entwickeln als proteinkodierende Gene.,
Es ist noch unklar, wie die Promoterevolution der Evolution von Menschen oder anderen höheren Organismen entsprechen könnte. Die Entwicklung eines Promotors, um aus einem bestimmten Genprodukt effektiv mehr oder weniger zu machen, ist jedoch eine faszinierende Alternative zur Entwicklung der Gene selbst.
Promotorstrukturen für RNA-Polymerasen I und III
Die Prozesse zum Einbringen von RNA-Polymerasen I und III in die DNA-Vorlage beinhalten etwas weniger komplexe Sammlungen von Transkriptionsfaktoren, aber das allgemeine Thema ist das gleiche.,
Die konservierten Promotorelemente für Gene, die von den Polymerasen I und III transkribiert werden, unterscheiden sich von denen, die von der RNA-Polymerase II transkribiert werden. RNA-Polymerase I transkribiert Gene, die zwei GC-reiche Promotorsequenzen in der Region -45 bis +20 aufweisen. Diese Sequenzen allein reichen aus, um die Transkriptionsinitiation durchzuführen, aber Promotoren mit zusätzlichen Sequenzen im Bereich von -180 bis -105 stromaufwärts der Initiationsstelle werden die Initiation weiter verbessern. Gene, die durch RNA-Polymerase III transkribiert werden, haben vorgeschaltete Promotoren oder Promotoren, die innerhalb der Gene selbst auftreten.,
Die eukaryotische Transkription ist ein streng regulierter Prozess, bei dem eine Vielzahl von Proteinen miteinander und mit dem DNA-Strang interagieren muss. Obwohl der Transkriptionsprozess bei Eukaryoten eine größere Stoffwechselinvestition erfordert als bei Prokaryoten, stellt er sicher, dass die Zelle genau die Prä-mRNAs transkribiert, die sie für die Proteinsynthese benötigt.,
Dehnung und Beendigung
Nach der Bildung des Preinitiationskomplexes wird die Polymerase von den anderen Transkriptionsfaktoren freigesetzt, und die Dehnung darf wie bei Prokaryoten mit der Polymerase fortfahren, die Pre-mRNA in der Richtung 5′ bis 3′ synthetisiert. Wie bereits diskutiert, transkribiert die RNA-Polymerase II den Hauptanteil der eukaryotischen Gene, daher konzentriert sich dieser Abschnitt darauf, wie diese Polymerase die Verlängerung und Beendigung erreicht.,
Obwohl der enzymatische Prozess der Dehnung bei Eukaryoten und Prokaryoten im Wesentlichen derselbe ist, ist die DNA-Schablone komplexer. Wenn sich eukaryotische Zellen nicht teilen, existieren ihre Gene als diffuse Masse von DNA und Proteinen, die Chromatin genannt werden. Die DNA ist in wiederholten Intervallen dicht um geladene Histonproteine verpackt. Diese DNA-Histon-Komplexe, zusammen Nukleosomen genannt, sind regelmäßig verteilt und umfassen 146 Nukleotide von DNA, die um acht Histone wie einen Faden um eine Spule gewickelt sind.,
Damit die Polynukleotidsynthese stattfinden kann, muss die Transkriptionsmaschine Histone jedes Mal aus dem Weg räumen, wenn sie auf ein Nukleosom trifft. Dies wird durch einen speziellen Proteinkomplex namens FACT erreicht, der für „erleichtert die Chromatin-Transkription.“Dieser Komplex zieht Histone von der DNA-Schablone weg, während sich die Polymerase entlang dieser bewegt. Sobald die Prä-mRNA synthetisiert ist, ersetzt der FACT-Komplex die Histone, um die Nukleosomen wiederherzustellen.
Die Beendigung der Transkription ist für die verschiedenen Polymerasen unterschiedlich., Anders als bei Prokaryoten erfolgt die Verlängerung durch RNA-Polymerase II in Eukaryoten 1.000-2.000 Nukleotide über das Ende des zu transkribierenden Gens hinaus. Dieser Prä-mRNA-Schwanz wird anschließend durch Spaltung während der mRNA-Verarbeitung entfernt. Andererseits erfordern die RNA-Polymerasen I und III Terminierungssignale. Gene, die von der RNA-Polymerase I transkribiert wurden, enthalten eine spezifische 18-Nukleotidsequenz, die von einem Terminierungsprotein erkannt wird. Der Prozess der Beendigung in der RNA-Polymerase III beinhaltet eine mRNA-Haarnadel ähnlich der rho-unabhängigen Beendigung der Transkription in Prokaryoten.,
Try It
Contribute!
Verbessern Sie diese Seitenlernen Sie mehr
- H Liang et al.“Fast evolution of core Promoter in Primaten-Genome“ Molecular Biology and Evolution 25 (2008): 1239-44. ↵















