Biologie voor Majors i
leerresultaten
- Beschrijf de rol van RNA-polymerase
Deze sectie zal de specifieke rol van RNA-polymerasen tijdens de transcriptie verder uitdiepen. Lees verder om de rol van de polymerases van RNA in elk stadium van transcriptie te leren.,
initiatie van transcriptie
In tegenstelling tot de prokaryotische polymerase die op zichzelf aan een DNA-sjabloon kan binden, hebben eukaryoten verschillende andere eiwitten nodig, transcriptiefactoren genaamd, om zich eerst aan het promotorgebied te binden en vervolgens de juiste polymerase te helpen werven.
de drie eukaryotische RNA-polymerasen
De kenmerken van eukaryotische mRNA-synthese zijn aanzienlijk complexer dan die van prokaryoten. In plaats van één enkele polymerase die vijf subeenheden omvat, hebben eukaryotes drie polymerasen die elk uit 10 subeenheden of meer worden samengesteld., Elke eukaryotic polymerase vereist ook een verschillende reeks transcriptiefactoren om het aan het malplaatje van DNA te brengen.
RNA-polymerase I bevindt zich in de nucleolus, een gespecialiseerde nucleaire substructuur waarin ribosomaal RNA (rRNA) wordt getranscribeerd, verwerkt en geassembleerd tot ribosomen (Tabel 1). De rRNA molecules worden beschouwd als structurele RNAs omdat zij een cellulaire rol hebben maar niet in proteã ne worden vertaald. RRNAs zijn componenten van het ribosoom en zijn essentieel voor het proces van vertaling. RNA-polymerase I synthetiseert alle rrnas behalve de 5S rRNA-molecule., De” S “aanduiding is van toepassing op” Svedberg ” eenheden, een niet-additieve waarde die de snelheid waarmee een deeltje sedimenten tijdens centrifugeren karakteriseert.
| Tabel 1.,f Transcription | α-Amanitin Sensitivity | |||
|---|---|---|---|---|
| I | Nucleolus | All rRNAs except 5S rRNA | Insensitive | |
| II | Nucleus | All protein-coding nuclear pre-mRNAs | Extremely sensitive | |
| III | Nucleus | 5S rRNA, tRNAs, and small nuclear RNAs | Moderately sensitive | |
RNA polymerase II is located in the nucleus and synthesizes all protein-coding nuclear pre-mRNAs., Eukaryotic pre-mRNAs ondergaan uitgebreide verwerking Na transcriptie maar vóór vertaling (figuur 1). Voor duidelijkheid, zal de bespreking van deze module van transcriptie en vertaling in eukaryotes de term “mRNAs” gebruiken om slechts de rijpe, verwerkte molecules te beschrijven die klaar zijn om te worden vertaald. De polymerase II van RNA is de oorzaak van het transcriberen van de overweldigende meerderheid van eukaryotic genen.

figuur 1. Eukaryotic mRNA bevat introns die moeten worden verbonden uit. Een 5 ‘cap en 3’ poly-a staart zijn ook toegevoegd.,
RNA-polymerase III bevindt zich ook in de kern. Deze polymerase transcribeert een verscheidenheid van structurele RNAs die 5S pre-rRNA, overdracht pre-RNAs (pre-tRNAs), en kleine nucleaire pre-RNAs omvat. TRNAs hebben een kritieke rol in vertaling; zij dienen als adaptermolecules tussen het mRNA-malplaatje en de groeiende polypeptideketen. Kleine nucleaire RNAs hebben een verscheidenheid van functies, met inbegrip van het “verbinden” pre-mRNAs en het regelen van transcriptiefactoren.,
een wetenschapper die een nieuw gen karakteriseert, kan bepalen welke polymerase het transcribeert door te testen of het gen wordt uitgedrukt in aanwezigheid van een bepaald paddenstoelengif, α-amanitine (Tabel 1). Interessant is dat α-amanitine, geproduceerd door Amanita phalloides, de paddenstoel des Doods Cap, de drie polymerasen zeer verschillend beïnvloedt. RNA-polymerase I is volledig ongevoelig voor α-amanitine, wat betekent dat de polymerase DNA in vitro kan transcriberen in aanwezigheid van dit gif. RNA-polymerase II is daarentegen extreem gevoelig voor α-amanitine en RNA-polymerase III is matig gevoelig., Het kennen van het transcriberen polymerase kan een onderzoeker in de algemene functie van het gen dat wordt bestudeerd aanwijzing. Omdat RNA-polymerase II de overgrote meerderheid van de genen transcribeert, zullen we ons op deze polymerase richten in onze daaropvolgende discussies over eukaryotische transcriptiefactoren en promotors.
RNA-Polymerase II-promotoren en transcriptiefactoren
eukaryotische promotoren zijn veel groter en complexer dan prokaryotische promotoren. Nochtans, hebben beide een opeenvolging gelijkend op de -10 opeenvolging van prokaryotes., In eukaryotes, wordt deze opeenvolging genoemd de doos van TATA, en heeft de consensusreeks TATAAA op de codeerstreng. Het bevindt zich op -25 tot -35 basen ten opzichte van de initiatie (+1) plaats (Figuur 2). Deze sequentie is niet identiek aan de E. coli -10 doos, maar het behoudt het A-T rijke element. De thermostabiliteit van A-T bindingen is laag en dit helpt het malplaatje van DNA om lokaal te ontspannen ter voorbereiding op transcriptie.,
in plaats van de eenvoudige σ-factor die helpt de prokaryotische RNA-polymerase aan zijn promotor te binden, assembleert eukaryotes een complex van transcriptiefactoren die worden vereist om RNA-polymerase II aan een eiwitcodagegen te rekruteren. Transcriptiefactoren die aan de promotor binden worden basale transcriptiefactoren genoemd. Deze basale factoren worden allemaal TFII (voor transcriptiefactor/polymerase II) plus een extra letter (A-J) genoemd. Het kerncomplex is TFIID, dat een Tata-bindende proteã ne (TBP) omvat., De andere transcriptiefactoren vallen systematisch op de DNA-template, waarbij elk het pre-initiatiecomplex verder stabiliseert en bijdraagt tot de rekrutering van RNA-polymerase II.
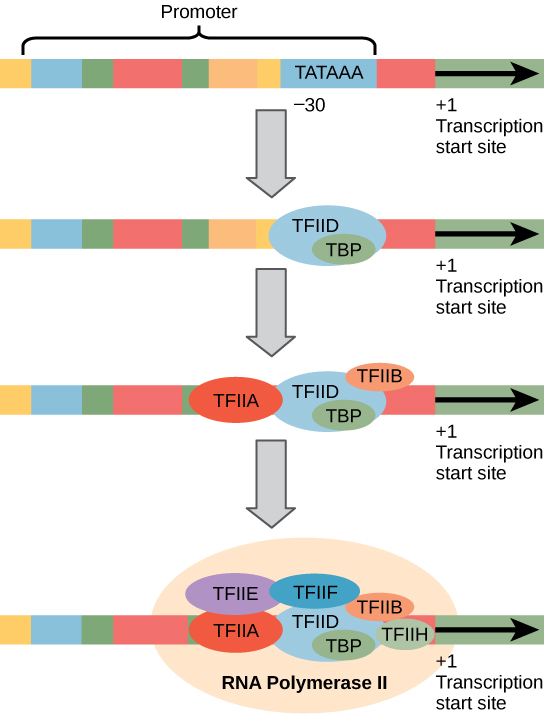
Figuur 2. Een gegeneraliseerde promotor van een gen getranscribeerd door RNA-polymerase II wordt getoond. Transcriptiefactoren herkennen de promotor. De polymerase II van RNA bindt dan en vormt de complexe transcriptieinitiatie.,
praktijkvraag
een wetenschapper splitst een eukaryotische promotor voor een bacterieel gen en voegt het gen in een bacterieel chromosoom. Verwacht je dat de bacterie het gen transcribeert?
sommige eukaryotische promotoren hebben ook een geconserveerde CAAT box (GGCCAATCT) bij ongeveer -80. Verder stroomopwaarts van de Tata box kunnen eukaryotische promotors ook een of meer GC-rijke dozen (GGCG) of octamer dozen (ATTTGCAT) bevatten., Deze elementen binden cellulaire factoren die de efficiency van transcriptieinitiatie verhogen en vaak in meer “actieve” genen worden geà dentificeerd die constant door de cel worden uitgedrukt.
basale transcriptiefactoren zijn cruciaal voor de vorming van een pre-initiatiecomplex op het DNA-sjabloon dat vervolgens RNA-polymerase II rekruteert voor transcriptie-initiatie. De complexiteit van eukaryotic transcriptie eindigt niet met de polymerases en promotors., Een leger van andere transcriptiefactoren, die aan stroomopwaartse versterkers en geluiddempers binden, helpt ook om de frequentie te regelen waarmee pre-mRNA van een gen wordt samengesteld. De versterkers en de geluiddempers beà nvloeden de efficiency van transcriptie maar zijn niet noodzakelijk voor transcriptie om te gaan.
de evolutie van promotors
De evolutie van genen kan een bekend begrip zijn. De veranderingen kunnen in genen tijdens de replicatie van DNA voorkomen, en het resultaat kan wel of niet voordelig voor de cel zijn., Door een enzym, een structureel eiwit of een andere factor te veranderen, kan het mutatieproces functies of fysieke kenmerken transformeren. Nochtans, kunnen eukaryotic promotors en andere gen regelgevende opeenvolgingen ook evolueren. Denk bijvoorbeeld aan een gen dat gedurende vele generaties waardevoller wordt voor de cel. Misschien codeert het gen een structureel eiwit dat de cel nodig heeft om in overvloed te synthetiseren voor een bepaalde functie. Als dit het geval is, zou het aan de cel voor de promotor van dat gen voordelig zijn om transcriptiefactoren efficiënter te werven en genuitdrukking te verhogen.,
wetenschappers die de evolutie van promotorsequenties onderzoeken, hebben verschillende resultaten gerapporteerd. Voor een deel, dit is omdat het moeilijk is om precies af te leiden waar een eukaryotic promotor begint en eindigt. Sommige promotors komen voor binnen genen; andere bevinden zich zeer ver stroomopwaarts, of zelfs stroomafwaarts, van de genen die zij reguleren. Nochtans, toen de onderzoekers hun onderzoek tot menselijke kernpromotor opeenvolgingen beperkten die experimenteel als opeenvolgingen werden gedefinieerd die het preinitiationcomplex binden, vonden zij dat de promotors zelfs sneller dan eiwit-codeert genen evolueren.,
Het is nog onduidelijk hoe de ontwikkeling van de promotor zou kunnen corresponderen met de evolutie van mensen of andere hogere organismen. Echter, de evolutie van een promotor om effectief min of meer van een bepaald genproduct te maken is een intrigerend alternatief voor de evolutie van de genen zelf.
Promotorstructuren voor RNA-polymerasen I en III
de processen om RNA-polymerasen I en III naar het DNA-sjabloon te brengen, hebben iets minder complexe verzamelingen van transcriptiefactoren, maar het algemene thema is hetzelfde.,
de geconserveerde promotorelementen voor genen getranscribeerd door polymerasen I en III verschillen van die getranscribeerd door RNA-polymerase II. RNA-polymerase I transcribeert genen die twee GC-rijke promotorsequenties hebben in het gebied -45 tot +20. Deze opeenvolgingen alleen zijn voldoende voor transcriptieinitiatie om voor te komen, maar de promotors met extra opeenvolgingen in het gebied van -180 aan -105 stroomopwaarts van de initiatieplaats zullen initiatie verder verbeteren. De genen die door de polymerase III van RNA worden getranscribeerd hebben stroomopwaartse promotors of promotors die binnen de genen zelf voorkomen.,
eukaryotische transcriptie is een strak geregeld proces dat een verscheidenheid van proteã nen vereist om met elkaar en met de bundel van DNA te interageren. Hoewel het proces van transcriptie in eukaryotes een grotere metabolische investering dan in prokaryotes impliceert, verzekert het dat de cel pre-mRNAs precies transcribeert dat het voor eiwitsynthese nodig heeft.,
elongatie en beëindiging
na de vorming van het pre-initiatiecomplex komt de polymerase vrij van de andere transcriptiefactoren, en de elongatie mag doorgaan zoals in prokaryoten met de polymerase die pre-mRNA synthetiseert in de 5′ tot 3′ – richting. Zoals eerder besproken, transcribeert de polymerase II van RNA het belangrijkste aandeel van eukaryotic genen, zodat zal deze sectie zich concentreren op hoe deze polymerase verlenging en beëindiging bereikt.,
hoewel het enzymatische proces van elongatie in wezen hetzelfde is in eukaryoten en prokaryoten, is het DNA-sjabloon complexer. Wanneer eukaryotic cellen niet delen, bestaan hun genen als diffuse massa van DNA en proteã nen genoemd chromatin. DNA is strak verpakt rond geladen histone proteã nen met herhaalde intervallen. Deze DNA-histoncomplexen, collectief nucleosomen genoemd, worden regelmatig uit elkaar geplaatst en omvatten 146 nucleotiden van DNA die rond acht histonen zoals draad rond een spoel worden gekronkeld.,
om polynucleotidesynthese tot stand te brengen, moeten de transcriptiemachines histonen uit de weg verplaatsen telkens wanneer het een nucleosoom tegenkomt. Dit wordt bereikt door een speciaal eiwitcomplex genoemd feit, dat Voor “vergemakkelijkt chromatin transcriptie staat.”Dit complex trekt histonen weg van het DNA-sjabloon als de polymerase langs het beweegt. Zodra pre-mRNA wordt samengesteld, vervangt het complexe feit de histonen om de nucleosomes opnieuw te maken.
De beëindiging van de transcriptie verschilt voor de verschillende polymerasen., In tegenstelling tot in prokaryotes, vindt de verlenging door de polymerase II van RNA in eukaryotes 1.000–2.000 nucleotiden voorbij het eind van het gen plaats dat wordt getranscribeerd. Deze pre-mRNA-staart wordt later verwijderd door splitsing tijdens mRNA-verwerking. Anderzijds, vereisen de polymerases I en III van RNA beëindigingssignalen. De genen die door de polymerase I van RNA worden getranscribeerd bevatten een specifieke 18-nucleotide opeenvolging die door een beëindigingsproteã ne wordt erkend. Het proces van beëindiging in de polymerase III van RNA impliceert een mRNA-haarspeld gelijkend op rho-onafhankelijke beëindiging van transcriptie in prokaryotes.,
probeer het
bijdragen!
verbeter deze pageleer meer
- H Liang et al.,” Fast evolution of core promotors in primate genomen, ” Molecular Biology and Evolution 25 (2008): 1239-44. ↵















