Anatomia polimerazy – jak funkcje i struktura są powiązane
dokładna replikacja genomu ma kluczowe znaczenie dla żywotności każdego organizmu. Ogólna koncepcja kopiowania DNA była widoczna po wyjaśnieniu podwójnej spiralnej struktury DNA i identyfikacji komplementarności pary zasad (1): jedna nić nukleobaz może służyć jako szablon do syntezy nowej nici. W ciągu dekady od tych odkryć, środek został oczyszczony z E. coli, który katalizuje powielanie nici DNA (2). Ten środek był określany jako „polimeraza”. E., polimeraza DNA coli I, pierwsza odkryta polimeraza DNA nie była pierwotną polimerazą replikacyjną, lecz zaangażowaną w opóźnioną rozdzielczość fragmentów Okazaki i naprawę DNA. To zapowiada przyszłe odkrycia wielu rodzin polimeraz DNA, z których każda służy specyficznym wymaganiom komórkowym do replikacji i naprawy DNA.
polimerazy DNA służą jako podstawowe enzymy w naukach przyrodniczych z tego samego powodu, że są krytyczne w naturze: kopiują DNA. Zastosowania polimerazy obejmują etykietowanie DNA, sekwencjonowanie i amplifikację., Jeden specyficzny protokół amplifikacji, reakcja łańcuchowa polimerazy (PCR) jest szeroko stosowaną techniką, która wykorzystuje termofilne polimerazy do wykładniczo amplifikacji określonych segmentów DNA (3) i umożliwia szereg zastosowań od diagnostyki człowieka i patogenu do klonowania molekularnego w laboratoriach biologicznych na całym świecie.
dokładność polimerazy
PCR stawia takie same podstawowe wymagania polimerazie, jak Komórka stawia system replikacji. Zasadniczo polimeraza powinna być niezawodna, dokładna i szybka., Dokładność polimerazy lub” wierność ” odnosi się do skłonności do włączenia WŁAŚCIWEGO nukleotydu, jak określono w nici wzorcowej. Standardowe enzymy PCR są, nic dziwnego, dość dokładne. Nawet polimeraza DNA Thermus aquaticus (Taq), uważana za polimerazę PCR o niskiej wierności, popełnia błąd tylko raz w przybliżeniu w insercjach nukleotydów (12)., Polimeraza odkrycia i inżynieria wysiłki produkowali high fidelity polymerase, które rzadko popełniają zasadowy substytucja błędy, wymagający DNA sekwencjonowanie metody czytać miliony zsyntetyzowanych zasad wykrywać wszelkie błędy przez polymerase postęp w mierzeniu wierności przez jednocząsteczkowe sekwencjonowanie zidentyfikowali Q5® High-Fidelity DNA Polymerase jako mający wierność 280X większa THANTAQ DNA polymerase (12).,
punkty kontrolne wierności: dobór geometryczny w miejscu aktywnym i poza nim
polimerazy DNA zapewniają dokładną replikację za pomocą szeregu molekularnych punktów kontrolnych, w miejscu inkorporacji nukleotydów i poza nim (1). Podczas dodawania nukleotydów, prawidłowy przychodzący nukleotyd jest pozycjonowany w celu produktywnego wyrównania grup katalitycznych, zapewniając skuteczne włączenie. To wyrównanie dla katalizy jest wrażliwe na zniekształcenia w położeniu spowodowane nieprawidłowym parowaniem bazy Watsona-Cricka, co pozwala na kinetyczne zwlekanie przy niepoprawnych lub nie poznanych parach bazy.,
korekta polimerazy DNA
inną metodą zwiększenia wierności jest aktywność egzonukleazy 3→5 polimerazy, określana jako „korekta”. Korzystając z opisanych powyżej punktów kontroli molekularnej bazowego parowania i aktywnego miejsca, polimeraza TAQ DNA jest niewiarygodnie dokładna, ale enzymy korekcyjne mogą mieć jeszcze większą wierność. Ta dodatkowa dokładność jest przekazywana poprzez korektę, z polimerazą „sprawdzającą”, czy właściwy nukleotyd został wprowadzony do szablonu., W przypadku wykrycia niedopasowania DNA jest przenoszone z domeny polimeryzacji do domeny egzonukleazy N-terminalnej 3→5 polimerazy. Nieprawidłowo wbudowany nukleotyd zostaje wycięty, DNA wraca do domeny polimeryzacji i kopiowanie może zostać wznowione (ryc. 2).
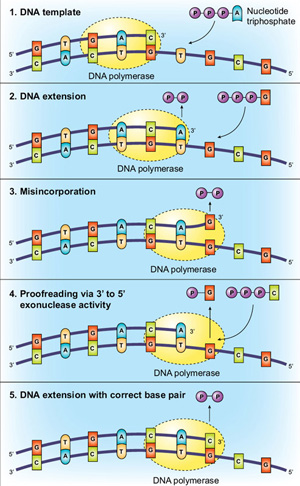
bakteriofag T4 okazał się użytecznym eksperymentalnym systemem do oceny znaczenia aktywności egzonukleazy 3→5 dla dokładnej replikacji DNA (6). Zidentyfikowano mutacje w genie T4 43 (kodującym polimerazę DNA), które zmniejszały lub zwiększały wierność. Definiując stosunek aktywności egzonukleazy/polimerazy (N/P) dla zmutowanego enzymu stwierdzono, że polimerazy o niskim stosunku N/P były bardziej podatne na błędy niż te o wysokim stosunku n / p., Wyjaśnienie tej obserwacji jest takie, że po włączeniu niedopasowanej zasady jest bardziej prawdopodobne, że egzonukleaza usunie nukleotyd, zanim aktywność polimerazy rozszerzy ją w enzymach o wyższych proporcjach N / P. Co ciekawe, skuteczność korekty polimerazy może wykazywać zależność sekwencji. Na przykład sekwencje bogate w AT są skuteczniej weryfikowane niż regiony GC. Uważa się, że ta rozbieżność wynika z niższej stabilności regionów AT, która ułatwia topienie nici, a tym samym aktywność korekty.,
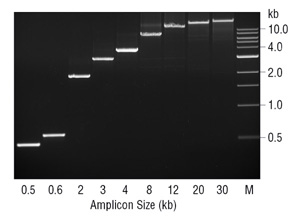
brak aktywności egzonukleazy 3→5 może mieć inne konsekwencje niż wierność w PCR. Zaproponowano brak aktywności korekcyjnej w Polimerazie DNA TAQ w celu ograniczenia możliwego rozmiaru amplikonu za pomocą tego enzymu (7). Ogólnie Taq działa najlepiej przy amplifikacji fragmentów DNA< 2 kb i może pracować z fragmentami do 3-4 kb. Przy zachowaniu tego rozmiaru amplikonu, Taq jest solidnym, łatwo zoptymalizowanym enzymem. Jednak powyżej ~3 kb szybko spada skuteczność., Podczas PCR, Taq będzie błędnie nukleotydy i produkować niedopasowania, w którym jest podatny na zwłokę i jest bardziej prawdopodobne dysocjacji przed rozszerzeniem w porównaniu do prawidłowo zasad parowane 3 końce. W związku z tym przy pewnej wielkości amplikonu i szybkości błędu polimerazy wystarczająca ilość niedopasowanych 3 końców może gromadzić się, aby skutecznie hamować proces PCR. Te niedopasowane końce 3 są szczególnie problematyczne dla Taq, ponieważ nie ma aktywności egzonukleazy 3→5, aby je usunąć., Poprzez dodanie niewielkiej ilości enzymu korekcyjnego, takiego jak polimeraza DNA Deep Vent®, można uzyskać amplifikację fragmentów ≥ 20 kb (ryc. 3). Ponieważ zdecydowaną większość enzymu w mieszance jest polimeraza TAQ DNA, prawdopodobnie wykonuje większość rozszerzenia podkładu, z korektą polimerazy głębokiego otworu wentylacyjnego, usuwając hamujące 3 niedopasowania generowane przez Taq.,
Processivity polimerazy
znaczenie korekty aktywności PCR jest powszechnie znany od prawie dwóch dekad, ale inna właściwość, processivity, zyskał większą uwagę., „Processivity” to termin, który odnosi się do liczby nukleotydów włączonych przez polimerazę w pojedynczym wiązaniu (przed dysocjacją). Polimeraza DNA TAQ dodaje około 50 nukleotydów na Wiązanie (8). Dlaczego to ma znaczenie? Polimeraza o niskiej procesywności lub” dystrybutywna ” rozszerza populację szablonów w zauważalnie inny sposób niż polimeraza procesywna. Wysoce dystrybutywna polimeraza wiąże się z szablonem, dodaje kilka nukleotydów i dysocjuje, pozostawiając populację szablonów, które mogą być równomiernie rozszerzone z czasem., Wysoce procesywna polimeraza wiąże szablon i rozszerza się z dłuższymi zdarzeniami wiązania.
wynikałoby z tego, że przy wystarczającej ilości czasu wynik reakcji polimerazy procesowej lub dystrybucyjnej byłby populacją skopiowanych szablonów. Jednak w pewnych okolicznościach możliwe jest, że polimeraza procesowa ma lepszą wydajność. Podjednostka α polimerazy E. coli, część głównej polimerazy replikacyjnej, ma procesowość < 10 par zasad i prędkość < 20 nukleotydów/sekundę (nt/s)., Jednakże, gdy podjednostka kojarzy się z innymi podjednostkami replisomu, w szczególności z zaciskiem przesuwnym, efektywna procesywność i szybkość replikacji zwiększają się do > odpowiednio 50 kb i 1000 nt/s (9). Termin „efektywna procesywność” jest używany, ponieważ istnieją dane wskazujące, że podjednostka polimerazy może wymieniać się w replisomie, ale replisom utrzymuje szybką, procesywną replikację DNA (10).
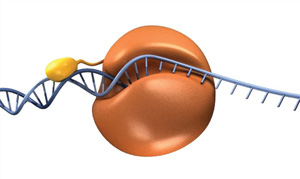
aby skorzystać z procesu w PCR, naukowcy połączyli domenę wiążącą DNA z archaealną polimerazą (11)., Ten chimeryczny enzym ma kilka ulepszonych właściwości, ale przede wszystkim jest w stanie wzmacniać DNA z krótszymi czasami rozszerzenia i produkować dłuższe produkty DNA bardziej efektywnie, skracając w ten sposób ogólny czas termocyklingu. Ten pomysł fuzji jest podstawą Q5 High-Fidelity DNA Polymerase i Phusion® High-Fidelity DNA Polymerase, dwóch polimeraz dostępnych z NEB (ryc. 4).
przyszłe kierunki
wiele właściwości wpływa na skuteczność i użyteczność polimerazy PCR. Architektura aktywnego miejsca polimerazy i aktywność korekty wpływają na dokładność produktu końcowego. Polimeraza łączy się i fuzja z białkiem wiążącym DNA, co zapewnia wyższą wydajność PCR pod względem długości amplikonu i, w przypadku Chimery, szybkości reakcji., Inne ważne postępy w PCR, tak jak Hot-start polimerazes wzrastać reakcja specyficzność, multipleks PCR (ryc. 5) i qPCR także zrewolucjonizowali Nauki przyrodnicze.
jak wykazały inżynierskie mieszanki i Chimery, właściwości samej polimerazy mogą być modulowane w celu poprawy wydajności PCR. W przyszłości jest prawdopodobne, że właściwości polimerazy będą coraz bardziej dostosowane do konkretnych zastosowań PCR, i jako takie, jest to ważny obszar badań w NEB.,

Zobacz wykres wyboru polimerazy DNA NEB















