Biologia dla kierunków i
efekty uczenia się
- opisz rolę polimerazy RNA
w tym rozdziale omówimy specyficzną rolę polimerazy RNA podczas transkrypcji. Czytaj dalej, aby poznać rolę polimeraz RNA na każdym etapie transkrypcji.,
inicjacja transkrypcji
w przeciwieństwie do prokariotycznej polimerazy, która sama może wiązać się z szablonem DNA, eukaryoty wymagają kilku innych białek, zwanych czynnikami transkrypcyjnymi, aby najpierw związać się z regionem promotora, a następnie pomóc w rekrutacji odpowiedniej polimerazy.
trzy eukariotyczne polimerazy RNA
cechy eukariotycznej syntezy mRNA są znacznie bardziej złożone niż prokariotyczne. Zamiast pojedynczej polimerazy składającej się z pięciu podjednostek, eukarioty mają trzy polimerazy, z których każda składa się z 10 podjednostek lub więcej., Każda eukariotyczna polimeraza wymaga również odrębnego zestawu czynników transkrypcyjnych, aby doprowadzić ją do szablonu DNA.
polimeraza RNA I znajduje się w jądrze, wyspecjalizowanej podbudowie jądrowej, w której rybosomalny RNA (rRNA) jest transkrybowany, przetwarzany i montowany w rybosomy (Tabela 1). Cząsteczki rRNA są uważane za strukturalne RNA, ponieważ pełnią rolę komórkową, ale nie są przekładane na białko. RRNA są składnikami rybosomu i są niezbędne w procesie translacji. Polimeraza RNA i syntetyzuje wszystkie rRNA z wyjątkiem cząsteczki 5s rRNA., Oznaczenie ” S „odnosi się do jednostek” Svedberga”, wartości nieaddytywnej, która charakteryzuje prędkość, z jaką cząstki osiadają podczas wirowania.
| Tabela 1.,f Transcription | α-Amanitin Sensitivity | |||
|---|---|---|---|---|
| I | Nucleolus | All rRNAs except 5S rRNA | Insensitive | |
| II | Nucleus | All protein-coding nuclear pre-mRNAs | Extremely sensitive | |
| III | Nucleus | 5S rRNA, tRNAs, and small nuclear RNAs | Moderately sensitive | |
RNA polymerase II is located in the nucleus and synthesizes all protein-coding nuclear pre-mRNAs., Eukariotyczne pre-mRNA poddawane są intensywnej obróbce po transkrypcji, ale przed translacją (ryc. 1). Dla jasności, w tym module omówienie transkrypcji i tłumaczenia u eukariotów użyje terminu „mRNA” do opisania tylko dojrzałych, przetworzonych cząsteczek, które są gotowe do tłumaczenia. Polimeraza RNA II odpowiada za transkrypcję przeważającej większości genów eukariotycznych.

Rysunek 1. Eukariotyczny mRNA zawiera introny, które muszą być rozdzielone. 5 'czapka i 3′ poly-a ogon są również dodawane.,
polimeraza RNA III znajduje się również w jądrze. Polimeraza ta transkrybuje różne strukturalne RNA, które zawierają 5s pre-rRNA, transfer Pre-RNA (pre-tRNA) i małe jądrowe pre-RNA. Trna mają krytyczną rolę w translacji; służą jako cząsteczki adaptera między szablonem mRNA a rosnącym łańcuchem polipeptydowym. Małe jądrowe RNA mają wiele funkcji, w tym „splicing” pre-mRNA i regulację czynników transkrypcyjnych.,
Naukowiec charakteryzujący nowy gen może określić, która polimeraza go transkrybuje, testując, czy gen jest wyrażony w obecności określonej trucizny grzybowej, α-amanityny (Tabela 1). Co ciekawe, α-amanityna wytwarzana przez Amanita phalloides, grzyb Czapki śmierci, wpływa na trzy polimerazy bardzo różnie. Polimeraza RNA I jest całkowicie niewrażliwa na α-amanitynę, co oznacza, że polimeraza może transkrybować DNA in vitro w obecności tej trucizny. W przeciwieństwie do tego polimeraza RNA II jest niezwykle wrażliwa na α-amanitynę, a polimeraza RNA III jest umiarkowanie wrażliwa., Znajomość transkrybującej polimerazy może podpowiedzieć badaczowi o ogólnej funkcji badanego genu. Ponieważ polimeraza RNA II transkrybuje zdecydowaną większość genów, skupimy się na tej polimerazie w kolejnych dyskusjach na temat eukariotycznych czynników transkrypcyjnych i promotorów.
promotory polimerazy RNA II i czynniki transkrypcyjne
promotory eukariotyczne są znacznie większe i bardziej skomplikowane niż promotory prokariotyczne. Jednak obie mają sekwencję podobną do -10 sekwencji prokariotów., U eukariotów sekwencja ta nazywana jest skrzynką TATA i ma ciąg konsensu TATAAA na nici kodującej. Znajduje się na podstawach od -25 do -35 w stosunku do miejsca inicjacji (+1) (ryc. 2). Sekwencja ta nie jest identyczna z pudełkiem E. coli -10, ale zachowuje pierwiastek bogaty w A-T. Termostabilność wiązań A-T jest niska, a to pomaga szablonowi DNA lokalnie odwijać się w przygotowaniu do transkrypcji.,
zamiast prostego czynnika σ, który pomaga związać prokariotyczną polimerazę RNA z jej promotorem, eukarioty tworzą kompleks czynników transkrypcyjnych wymaganych do rekrutacji polimerazy RNA II do genu kodującego białko. Czynniki transkrypcyjne, które wiążą się z promotorem, nazywane są podstawnymi czynnikami transkrypcyjnymi. Wszystkie te podstawowe czynniki nazywane są TFII (dla czynnika transkrypcyjnego/polimerazy II) plus dodatkowa litera (A-J). Głównym kompleksem jest TFIID, który zawiera białko wiążące TATA (TBP)., Pozostałe czynniki transkrypcyjne systematycznie ulegają rozkładowi na szablonie DNA, z których każdy dodatkowo stabilizuje kompleks przed inicjacją i przyczynia się do rekrutacji polimerazy RNA II.
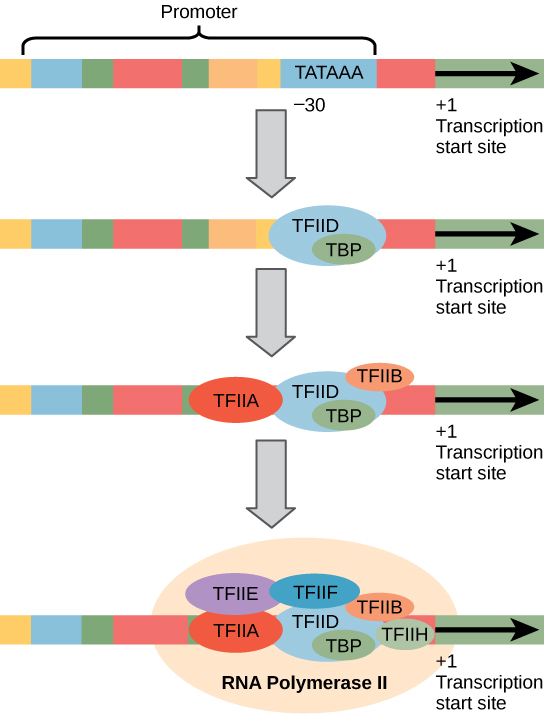
Rysunek 2. Przedstawiono uogólniony promotor genu transkrybowanego przez polimerazę RNA II. Czynniki transkrypcyjne rozpoznają promotora. Polimeraza RNA II wiąże się następnie i tworzy kompleks inicjacji transkrypcji.,
pytanie praktyczne
Naukowiec łączy promotor eukariotyczny przed genem bakteryjnym i wstawia Gen do chromosomu bakteryjnego. Spodziewasz się, że bakterie transkrypują Gen?
niektóre promotory eukariotyczne mają również zachowaną skrzynkę CAAT (GGCCAATCT) w przybliżeniu -80. W dalszej kolejności od pola TATA promotory eukariotyczne mogą również zawierać jedno lub więcej pól bogatych w GC (GGCG) lub oktamerów (ATTTGCAT)., Elementy te wiążą czynniki komórkowe, które zwiększają efektywność inicjacji transkrypcji i są często identyfikowane w bardziej” aktywnych ” genach, które są stale wyrażane przez komórkę.
podstawowe czynniki transkrypcyjne są kluczowe w tworzeniu kompleksu preinicjacyjnego na szablonie DNA, który następnie rekrutuje polimerazę RNA II do inicjacji transkrypcji. Złożoność transkrypcji eukariotycznej nie kończy się na polimerazach i promotorach., Armia innych czynników transkrypcyjnych, które wiążą się z wzmacniaczami i tłumikami, również pomaga regulować częstotliwość, z jaką pre-mRNA jest syntetyzowany z genu. Wzmacniacze i tłumiki wpływają na efektywność transkrypcji, ale nie są konieczne do transkrypcji.
Ewolucja promotorów
ewolucja genów może być znanym pojęciem. Mutacje mogą wystąpić w genach podczas replikacji DNA, a wynik może lub nie może być korzystny dla komórki., Poprzez zmianę enzymu, białka strukturalnego lub innego czynnika, proces mutacji może przekształcić funkcje lub cechy fizyczne. Jednak promotory eukariotyczne i inne sekwencje regulacyjne genów mogą również ewoluować. Na przykład rozważ gen, który przez wiele pokoleń staje się bardziej wartościowy dla komórki. Może Gen koduje białko strukturalne, które komórka potrzebuje syntetyzować w obfitości dla określonej funkcji. W takim przypadku korzystne byłoby dla komórki, aby promotor tego genu rekrutował czynniki transkrypcyjne bardziej efektywnie i zwiększał ekspresję genu.,
naukowcy badający ewolucję sekwencji promotorów zgłaszali różne wyniki. Po części jest tak, ponieważ trudno jest dokładnie wywnioskować, gdzie zaczyna się i kończy eukariotyczny promotor. Niektóre promotory występują w obrębie genów; inne znajdują się bardzo daleko w górę, lub nawet w dół, genów, które regulują. Jednak kiedy naukowcy ograniczyli swoje badania do sekwencji promotorów rdzenia ludzkiego, które zostały zdefiniowane eksperymentalnie jako sekwencje wiążące kompleks preinicjacyjny, odkryli, że promotory ewoluują jeszcze szybciej niż geny kodujące białka.,
nadal nie jest jasne, w jaki sposób ewolucja promotora może odpowiadać ewolucji ludzi lub innych organizmów wyższych. Jednak ewolucja promotora, aby skutecznie wytwarzać więcej lub mniej danego produktu genowego, jest intrygującą alternatywą dla ewolucji samych genów.
struktury Promotorowe polimeraz RNA I I III
procesy wprowadzania polimeraz RNA i I III do szablonu DNA obejmują nieco mniej złożone zbiory czynników transkrypcyjnych, ale ogólny temat jest taki sam.,
zachowane elementy promotora dla genów transkrybowanych przez polimerazy i I III różnią się od tych transkrybowanych przez polimerazę RNA II. polimeraza RNA i transkrybuje geny, które mają dwie sekwencje promotora bogate w GC w regionie -45 do +20. Same sekwencje są wystarczające do rozpoczęcia transkrypcji, ale promotory z dodatkowymi sekwencjami w regionie od -180 do -105 przed miejscem inicjacji będą jeszcze bardziej nasilać inicjację. Geny transkrybowane przez polimerazę RNA III mają promotory lub promotory występujące w samych genach.,
transkrypcja eukariotyczna jest ściśle regulowanym procesem, który wymaga różnych białek do interakcji ze sobą i z nicią DNA. Chociaż proces transkrypcji u eukariotów wymaga większych inwestycji metabolicznych niż u prokariotów, zapewnia to, że komórka transkrybuje dokładnie pre-mRNA, których potrzebuje do syntezy białek.,
Wydłużenie i zakończenie
Po utworzeniu kompleksu preinicjacyjnego polimeraza jest uwalniana z innych czynników transkrypcyjnych, a wydłużenie może przebiegać tak, jak w prokariotach, z polimerazą syntetyzującą pre-mRNA w kierunku 5′ do 3′. Jak wspomniano wcześniej, polimeraza RNA II transkrybuje główny udział genów eukariotycznych, więc ta sekcja skupi się na tym, jak polimeraza osiąga wydłużenie i zakończenie.,
chociaż enzymatyczny proces elongacji jest zasadniczo taki sam u eukariotów i prokariotów, szablon DNA jest bardziej złożony. Gdy komórki eukariotyczne nie dzielą się, ich geny występują jako rozproszona masa DNA i białek zwana chromatyną. DNA jest szczelnie pakowane wokół naładowanych białek histonowych w powtarzających się odstępach czasu. Te kompleksy DNA-histonu, zwane łącznie nukleosomami, są regularnie rozmieszczone i zawierają 146 nukleotydów DNA rany wokół ośmiu histonów jak nici wokół szpuli.,
aby doszło do syntezy polinukleotydów, maszyna transkrypcyjna musi usunąć histony z drogi za każdym razem, gdy napotka nukleosom. Osiąga się to dzięki specjalnemu kompleksowi białkowemu o nazwie FACT, który oznacza ” ułatwia transkrypcję chromatyny.”Ten kompleks odciąga histony od szablonu DNA, gdy polimeraza porusza się po nim. Po zsyntetyzowaniu pre-mRNA kompleks FACT zastępuje histony w celu odtworzenia nukleosomów.
zakończenie transkrypcji jest różne dla różnych polimeraz., W przeciwieństwie do prokariotów, elongacja przez polimerazę RNA II u eukariotów odbywa się 1000–2000 nukleotydów poza końcem transkrypcji genu. Ten pre-mRNA ogon jest następnie usuwany przez rozcięcie podczas przetwarzania mRNA. Z drugiej strony polimerazy RNA I I III wymagają sygnałów zakończenia. Geny transkrybowane przez polimerazę RNA I zawierają specyficzną 18-nukleotydową sekwencję, która jest rozpoznawana przez białko terminacyjne. Proces terminacji w polimerazie RNA III obejmuje spinkę mRNA podobną do Rho-niezależnego zakończenia transkrypcji u prokariotów.,
Try It
Contribute!
popraw tę stronęucz się więcej
- H Liang et al., „Fast evolution of core promoters in primate genomes,” Molecular Biology and Evolution 25 (2008): 1239-44. ↵















