Anatomi af en Polymerase – Hvordan Funktion og Struktur er Relateret
Præcis genom replikation er afgørende for levedygtigheden af enhver organisme. Det generelle koncept for kopiering af DNA var tydeligt ved opklaringen af DNA ‘ s dobbelt-spiralformet struktur og identifikation af basepar komplementaritet (1): en streng af nucleobases kunne tjene som skabelon for syntesen af en ny strand. Inden for et årti efter disse opdagelser blev et middel renset fra E. coli, der katalyserede DNA-strengduplikation (2). Dette middel blev betegnet som en”polymerase”. E., coli-DNA-Polymerase jeg, den første DNA-polymerase, der opdagede, var ikke det primære-polymerase, replikation, men i stedet én, der er involveret i tilbagestående strand Okazaki-fragment opløsning og DNA-reparation. Dette foreshado .ed fremtidige opdagelser af mange DNA-polymerasefamilier, der hver tjener specifikke cellulære krav til DNA-replikation og reparation.
DNA-polymeraser tjener som grundlæggende en .ymer i biovidenskaben af samme grund som de er kritiske: de kopierer DNA. Polymerase applikationer omfatter DNA-mærkning, sekventering og amplifikation., En bestemt forstærkning protokol, polymerase chain reaction (PCR) er en meget anvendt teknik, der anvender termofile polymeraser til eksponentielt forstærke specifikke DNA-segmenter (3) og giver en vifte af applikationer fra human-og patogen diagnostik molekylær kloning af mennesker, biologi laboratorier rundt om i verden.
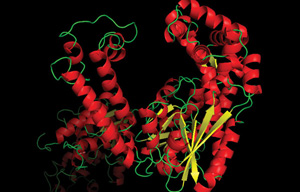
– Polymerase Nøjagtighed
PCR sætter de samme grundlæggende krav om en polymerase som en celle sætter på sin replikation system. I det væsentlige bør polymerasen være pålidelig, præcis og hurtig., Polymerasenøjagtighed eller “troskab” henviser til tilbøjelighed til at inkorporere det korrekte nukleotid som specificeret af skabelonstrengen. Standard PCR-en .ymerne er ikke overraskende ret præcise. Selv Thermus a .uaticus (Ta.) DNA-polymerase, der betragtes som en lav fidelity PCR-polymerase, begår kun en fejl en gang i cirka nukleotidindsættelser (12)., Polymerase opdagelse og teknik indsats har produceret high fidelity-polymeraser, som sjældent laver base substitution fejl, som kræver, at DNA-sekventering metoder til at læse millioner af syntetiseret baser til at opdage eventuelle fejl ved hjælp af polymerase Fremskridt i måling troskab ved enkelt-molekyle sekventering har identificeret Q5® High-Fidelity-DNA-Polymerase, som der troskab 280X større thanTaq DNA-polymerase (12).,
Fidelity checkpoints: geometrisk udvælgelse på det aktive sted og videre
DNA-polymeraser sikrer nøjagtig replikation ved hjælp af en række molekylære checkpoints, på stedet for nukleotidindtagelse og videre (1). Under nukleotidaddition er det korrekte indkommende nukleotid placeret til en produktiv justering af katalytiske grupper, hvilket sikrer effektiv inkorporering. Denne justering til katalyse er følsom over for forvrængninger i position forårsaget af forkert baseatson-Crick-baseparring, hvilket giver mulighed for kinetisk standsning ved forkerte eller ikke-beslægtede basepar.,
Korrekturlæsning af DNA-Polymeraser
en Anden metode til at øge troskab er for polymerase at have 3→5 exonuclease aktivitet, som kan kaldes “korrekturlæsning”. Ved hjælp af baseparring og aktive molekylære kontrolpunkter beskrevet ovenfor er Ta.DNA-polymerase utroligt nøjagtig, men korrekturlæsningsen .ymer kan have endnu højere troskab. Denne yderligere nøjagtighed formidles via korrekturlæsning, hvor polymerasen “kontrollerer”, om det korrekte nukleotid er indsat i skabelonen., Hvis der påvises en uoverensstemmelse, overføres DNA ‘ et fra polymerisationsdomænet til et N-terminal 3 5 5 e .onuclease-domæne af polymerasen. Det forkert inkorporerede nukleotid udskæres, DNA bevæger sig tilbage i polymerisationsdomænet, og kopiering kan genoptages (figur 2).
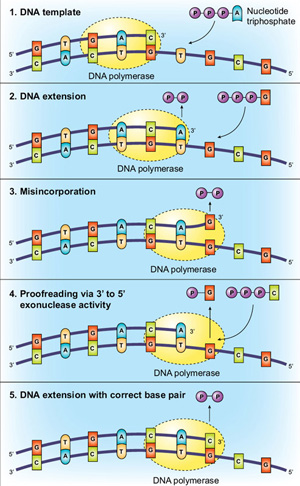
Bakteriofag T4 vist sig at være et nyttigt eksperimentelt system til evaluering af betydningen af, at 3→5 exonuclease aktivitet for præcis DNA-replikation (6). Mutationer i T4 gen 43 (som koder for DNA-polymerasen) blev identificeret, der enten faldt eller øgede troskab. Ved at definere et e .onuclease/polymerase (N/p) aktivitetsforhold for et mutant en .ym blev det konstateret, at polymeraser med lave n/p-forhold var mere udsatte for fejl end dem med høje n / p-forhold., En forklaring på denne observation er, at ved indførelse af et forkert udgangspunkt, at det er mere sandsynligt, at exonuclease vil fjerne nukleotid før polymerase aktivitet omfatter det i enzymer med højere N/P-forhold. Interessant nok kan korrekturlæsningseffektiviteten af en polymerase vise sekvensafhængighed. For eksempel er at-rige sekvenser mere effektivt korrekturlæst end GC-regioner. Denne uoverensstemmelse menes at skyldes den lavere stabilitet i AT-regioner, der letter strengsmeltning og derfor korrekturlæsningsaktivitet.,
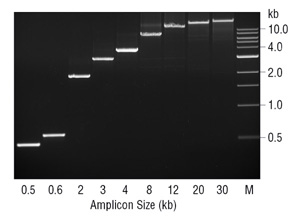
fraværet af 3 5 5 e .onuclease-aktivitet kan have andre forgreninger end troskab i PCR. Manglen på korrekturlæsningsaktivitet i Ta.DNA-Polymerase er blevet foreslået for at begrænse den mulige ampliconstørrelse med dette en .ym (7). Generelt fungerer Ta.bedst, når man forstærker DNA-fragmenter < 2 kb, og kan arbejde med fragmenter op til 3-4 kb. Når den holdes til denne ampliconstørrelse, er Ta.et robust, let optimeret en .ym. Men over ~3 kb falder det hurtigt i effektivitet., Under PCR, Ta.vil misincorporere nukleotider og producere uoverensstemmelser, hvor den er tilbøjelig til at stoppe og er mere tilbøjelig til at dissociere, før den strækker sig sammenlignet med korrekt baseparret 3 ender. Derfor kan der ved en bestemt ampliconstørrelse og polymerasefejl akkumuleres nok uoverensstemmende 3 ender til effektivt at hæmme PCR-processen. Disse uoverensstemmende 3 ender er særligt problematiske for Ta., fordi det mangler 3 5 5 e .onuclease-aktiviteten for at fjerne dem., Ved tilsætning i en lille mængde korrekturlæsningsen .ym, såsom Deep Vent DNA DNA-Polymerase, kan amplifikation af fragmenter 20 20 kb opnås (figur 3). Da langt størstedelen af en .ymet i blandingen er Ta.DNA-Polymerase, gør det sandsynligvis hovedparten af primerforlængelsen, idet korrekturlæsningsdybdepolymerasen fjerner de hæmmende 3-uoverensstemmelser genereret af Ta..,
– Polymerase Processivity
betydningen af korrekturlæsning aktivitet PCR, har været kendt i næsten to årtier, men en anden ejendom, processivity, har fået øget opmærksomhed., “Processivitet” er et udtryk, der refererer til antallet af nukleotider inkorporeret af en polymerase i en enkelt bindende begivenhed (før dissociation). 50 nukleotider pr. bindingsbegivenhed (8). Hvorfor betyder det noget? En lavprocessivitet eller “distributiv” polymerase udvider en population af skabeloner på en mærkbar anden måde end en processiv polymerase. En stærkt distributiv polymerase binder til en skabelon, tilføjer et par nukleotider og dissocierer, hvilket efterlader en population af skabeloner, der kan udvides lige med tiden., En meget processiv polymerase binder en skabelon og strækker sig med længere bindende begivenheder.
det ville følge, at givet tid nok resultatet af enten en processiv eller distributiv polymerase reaktion ville være en population af kopierede skabeloner. Under visse omstændigheder er det imidlertid muligt, at den processive polymerase har overlegen ydelse. E. coli-polymerase III α-subunit, en del af main-polymerase, replikation, har en processivity af < 10 base-par og en hastighed < 20 nukleotider/andet (nt/s)., Men når subunit associerede virksomheder med de andre replisome underenheder, især de glidende klemme, effektiv processivity og replikation hastighed stige til > 50 kb 1,000 nt/s, henholdsvis (9). Udtrykket “effektiv processivitet” bruges, fordi der er data, der indikerer, at polymeraseunderenheden kan udveksle i replisomet, men replisomet opretholder hurtig, processiv DNA-replikation (10).
for at drage fordel af processivitet i PCR har forskere smeltet et DNA-bindingsdomæne til en arkaeal polymerase (11)., Dette kimære enzym har flere forbedrede egenskaber, men især det, er i stand til at forstærke DNA med kortere forlængelse gange og producere længere DNA-produkter mere effektivt og dermed forkorte den samlede thermocycling gange. Denne fusionsidee er grundlaget for55 High-Fidelity DNA Polymerase og Phusion DNA High-Fidelity DNA Polymerase, to polymeraser tilgængelige fra NEB (figur 4).
fremtidige retninger
mange egenskaber påvirker effektiviteten og anvendeligheden af en PCR-polymerase. Polymerase aktiv site arkitektur og korrekturlæsning aktivitet påvirker nøjagtigheden af det endelige produkt. Polymeraseblandinger og fusion til et DNA-bindende protein giver overlegen PCR-ydeevne for ampliconlængde og, for Kimærernes vedkommende, reaktionshastighed., Andre vigtige fremskridt inden for PCR, såsom Hot-start-polymeraser for at øge reaktionsspecificitet, multiple. – PCR (figur 5) og .pcr har også revolutioneret biovidenskaberne.
som påvist ved manipulerede blandinger og kimærer, egenskaber af polymerasen selv kan moduleres til at forbedre PCR-ydeevne. I fremtiden er det sandsynligt, at polymeraseegenskaber i stigende grad vil blive skræddersyet til specifikke PCR-applikationer, og som sådan er dette et vigtigt forskningsområde hos neb.,

Vis Nebs DNA-Polymerase-Udvælgelsesdiagram















