Anatomía de una polimerasa: cómo se relacionan la función y la estructura
la replicación precisa del genoma es crítica para la viabilidad de cualquier organismo. El concepto general para copiar ADN fue evidente sobre la elucidación de la estructura doble helicoidal del ADN y la identificación de la complementariedad de pares de Bases (1): una hebra de nucleobases podría servir como la plantilla para la síntesis de una nueva hebra. Dentro de una década de estos descubrimientos, un agente fue purificado de E. coli que catalizó la duplicación de cadenas de ADN (2). Este agente fue denominado una «polimerasa». E., la polimerasa de ADN coli i, la primera polimerasa de ADN descubierta no fue la polimerasa replicativa primaria, sino que participó en la resolución de fragmentos de Okazaki y la reparación del ADN. Esto prefiguró futuros descubrimientos de muchas familias de ADN polimerasa, cada una sirviendo a requerimientos celulares específicos para la replicación y reparación del ADN.
Las ADN polimerasas sirven como enzimas fundamentales en las ciencias de la vida por la misma razón que son de naturaleza crítica: copian el ADN. Las aplicaciones de la polimerasa incluyen el etiquetado de ADN, la secuenciación y la amplificación., Un protocolo de amplificación específico, la reacción en cadena de la polimerasa (PCR) es una técnica ampliamente utilizada que emplea polimerasas termofílicas para amplificar exponencialmente segmentos específicos de ADN (3) y permite una gama de aplicaciones desde el diagnóstico de humanos y patógenos hasta la clonación molecular en laboratorios de biología de todo el mundo.
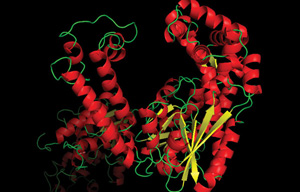
de la Polimerasa Precisión
PCR pone las mismas exigencias básicas en una polimerasa como una célula se pone en su sistema de replicación. Esencialmente, la polimerasa debe ser confiable, precisa y rápida., La precisión de la polimerasa o» fidelidad » se refiere a la propensión a incorporar el nucleótido correcto según lo especificado por la hebra de plantilla. Las enzimas PCR estándar son, como era de esperar, bastante precisas. Incluso la polimerasa de ADN Thermus aquaticus (Taq), considerada una polimerasa PCR de baja fidelidad, solo comete un error una vez en aproximadamente inserciones de nucleótidos (12)., El descubrimiento de la polimerasa y los esfuerzos de ingeniería han producido polimerasas de alta fidelidad, que rara vez cometen errores de sustitución de bases, requiriendo métodos de secuenciación de ADN para leer millones de bases sintetizadas para detectar cualquier error por la polimerasa.los avances en la medición de la fidelidad por secuenciación de una sola molécula han identificado a la polimerasa de ADN de alta fidelidad Q5® con una fidelidad 280 veces mayor que la polimerasa de ADN de thanTaq (12).,
puntos de control de fidelidad: la selección geométrica en el sitio activo y más allá
las polimerasas de ADN garantizan una replicación precisa utilizando una serie de puntos de control moleculares, en el sitio de incorporación de nucleótidos y más allá (1). Durante la adición de nucleótidos, el nucleótido entrante correcto se posiciona para una alineación productiva de los grupos catalíticos, asegurando una incorporación eficiente. Esta alineación para la catálisis es sensible a las distorsiones en la posición causadas por el emparejamiento incorrecto de la base Watson-Crick, lo que permite el estancamiento cinético en pares de bases incorrectos o no afines.,
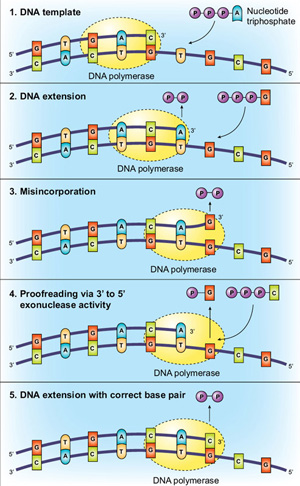
corrección de ADN polimerasas
otro método para aumentar la fidelidad es que la polimerasa tenga una actividad de exonucleasa de 3→5, denominada «corrección de pruebas». Usando el emparejamiento de bases y los puntos de control moleculares del sitio activo descritos anteriormente, la polimerasa de ADN Taq es increíblemente precisa, pero las enzimas de revisión pueden tener una fidelidad aún mayor. Esta precisión adicional se transmite a través de la revisión, con la polimerasa «verificando» si el nucleótido correcto se ha insertado en la plantilla., Si se detecta un desajuste, el ADN se transfiere del dominio de polimerización a un dominio de exonucleasa 3→5 N-terminal de la polimerasa. El nucleótido incorporado incorrectamente es extirpado, el ADN regresa al dominio de polimerización y se puede reanudar la copia (Figura 2).
el bacteriófago T4 demostró ser un sistema experimental útil para evaluar la importancia de la actividad de exonucleasa 3→5 para la replicación precisa del ADN (6). Se identificaron mutaciones en el gen T4 43 (que codifica la ADN polimerasa) que disminuyeron o aumentaron la fidelidad. Al definir una relación de actividad exonucleasa / polimerasa (n / P) para una enzima mutante, se encontró que las polimerasas con relaciones n/P bajas eran más propensas a errores que aquellas con relaciones n/P altas., Una explicación para esta observación es que al incorporar una base no coincidente es más probable que la exonucleasa elimine el nucleótido antes de que la actividad de la polimerasa lo extienda en enzimas con relaciones n/P más altas. Curiosamente, la eficacia de corrección de una polimerasa puede mostrar dependencia de secuencia. Por ejemplo, las secuencias AT-rich son corregidas de manera más efectiva que las regiones GC. Se cree que esta discrepancia se debe a la menor estabilidad de las regiones AT que facilita la fusión de la cadena y, por lo tanto, la actividad de revisión.,
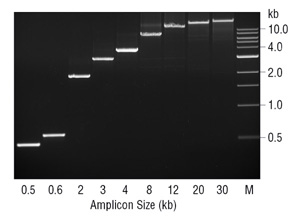
la ausencia de actividad de exonucleasa 3→5 puede tener ramificaciones distintas a la fidelidad en la PCR. La falta de actividad de revisión en la polimerasa de ADN Taq se ha propuesto para limitar el tamaño del amplicón posible con esta enzima (7). Generalmente, Taq funciona mejor cuando amplifica fragmentos de ADN < 2 kb, y puede trabajar con fragmentos de hasta 3-4 kb. Cuando se mantiene este tamaño de amplicón, Taq es una enzima robusta y fácilmente optimizada. Sin embargo, por encima de ~3 kb cae rápidamente en efectividad., Durante la PCR, Taq incorporará incorrectamente nucleótidos y producirá desajustes, en los que es propenso a estancarse y es más probable que se disocie antes de extenderse en comparación con la base emparejada correctamente 3 extremos. Por lo tanto, a un cierto tamaño de amplicón y una tasa de error de polimerasa, se pueden acumular suficientes extremos no coincidentes para inhibir efectivamente el proceso de PCR. Estos 3 extremos no coincidentes son particularmente problemáticos para Taq porque carece de la actividad de exonucleasa 3→5 para eliminarlos., Mediante la adición de una pequeña cantidad de enzima de revisión, como la ADN polimerasa Deep Vent®, se puede lograr una amplificación de fragmentos ≥ 20 kb (Figura 3). Dado que la gran mayoría de la enzima en la mezcla es la polimerasa de ADN Taq, probablemente esté haciendo la mayor parte de la extensión de imprimación, con la corrección de la polimerasa de ventilación profunda eliminando los desajustes inhibitorios 3 generados por Taq.,
Processivity de la polimerasa
la importancia de la actividad de corrección de pruebas para PCR ha sido ampliamente conocida durante casi dos décadas, pero otra propiedad, la processivity, ha ganado mayor atención., «Procesividad» es un término que se refiere al número de nucleótidos incorporados por una polimerasa en un solo evento de unión (antes de la disociación). La ADN polimerasa Taq agrega aproximadamente 50 nucleótidos por evento de unión (8). ¿Por qué importa esto? Una polimerasa de baja processividad o «distributiva» extiende una población de plantillas de una manera notablemente diferente a una polimerasa procesiva. Una polimerasa altamente distributiva se une a una plantilla, agrega un par de nucleótidos y se disocia, dejando una población de plantillas que se puede extender igualmente con el tiempo., Una polimerasa altamente procesiva se une a una plantilla y se extiende con eventos de unión más largos.
se deduce que, dado el tiempo suficiente, el resultado de una reacción de polimerasa procesiva o distributiva sería una población de plantillas copiadas. Sin embargo, en ciertas circunstancias es posible que la polimerasa procesiva tenga un rendimiento superior. La subunidad α de la polimerasa III de E. coli, parte de la polimerasa replicativa principal, tiene una processividad de < 10 pares de bases y una velocidad de < 20 nucleótidos/segundo (nt/s)., Sin embargo, cuando la subunidad se asocia con las otras subunidades de replisoma, particularmente la abrazadera deslizante, la processividad efectiva y la velocidad de replicación aumentan a > 50 kb y 1,000 nt/s, respectivamente (9). El término «processividad efectiva» se usa porque hay datos que indican que la subunidad de la polimerasa puede intercambiarse en el replisoma, pero el replisoma mantiene una replicación rápida y procesiva del ADN (10).
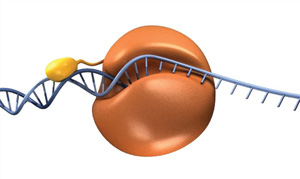
para aprovechar la processividad en la PCR, los investigadores han fusionado un dominio de unión de ADN a una polimerasa arqueal (11)., Esta enzima quimérica tiene varias propiedades mejoradas, pero notablemente es capaz de amplificar el ADN con tiempos de extensión más cortos y producir productos de ADN Más largos de manera más eficiente, acortando así los tiempos generales de termociclado. Esta idea de fusión es la base de la ADN polimerasa de alta fidelidad Q5 y la ADN polimerasa de alta fidelidad Phusion®, dos polimerasas disponibles en NEB (Figura 4).
direcciones futuras
muchas propiedades afectan la eficacia y utilidad de una polimerasa PCR. La arquitectura del sitio activo de la polimerasa y la actividad de revisión afectan la precisión del producto final. Las mezclas de polimerasa y la fusión a una proteína de unión al ADN confieren un rendimiento de PCR superior para la longitud del amplicón y, en el caso de la Quimera, la velocidad de reacción., Otros avances importantes en la PCR, como las polimerasas de arranque en caliente para aumentar la especificidad de la reacción, la PCR múltiple (Figura 5) y la qPCR también han revolucionado las ciencias de la vida.
como lo demuestran las mezclas de ingeniería y las quimeras, las propiedades de la propia polimerasa pueden ser moduladas para mejorar el rendimiento de la PCR. En el futuro, es probable que las propiedades de la polimerasa se adapten cada vez más a aplicaciones específicas de PCR, y como tal, este es un área importante de investigación en NEB.,

Ver Tabla de selección de ADN polimerasa de NEB















