Anatomia di una polimerasi – Come funzione e struttura sono correlati
La replica accurata del genoma è fondamentale per la vitalità di qualsiasi organismo. Il concetto generale per la copia del DNA era evidente dopo la delucidazione della struttura a doppia elica del DNA e l’identificazione della complementarità della coppia di basi (1): un filamento di nucleobasi poteva servire da modello per la sintesi di un nuovo filamento. Entro un decennio da queste scoperte, un agente è stato purificato da E. coli che ha catalizzato la duplicazione del filamento di DNA (2). Questo agente è stato definito una “polimerasi”. E., coli DNA polimerasi I, la prima DNA polimerasi scoperto non era la polimerasi replicativa primaria, ma invece uno coinvolto nella risoluzione del frammento di Okazaki filamento in ritardo e riparazione del DNA. Ciò prefigurava future scoperte di molte famiglie di DNA polimerasi, ognuna delle quali rispondeva a specifici requisiti cellulari per la replicazione e la riparazione del DNA.
Le DNA polimerasi servono come enzimi fondamentali nelle scienze della vita per la stessa ragione per cui sono di natura critica: copiano il DNA. Le applicazioni della polimerasi includono l’etichettatura, il sequenziamento e l’amplificazione del DNA., Uno specifico protocollo di amplificazione, la reazione a catena della polimerasi (PCR) è una tecnica ampiamente utilizzata che impiega polimerasi termofile per amplificare esponenzialmente specifici segmenti di DNA (3) e consente una gamma di applicazioni dalla diagnostica umana e patogeno alla clonazione molecolare nei laboratori di biologia di tutto il mondo.
Precisione della polimerasi
La PCR pone le stesse esigenze di base su una polimerasi come una cella mette sul suo sistema di replica. Essenzialmente, la polimerasi dovrebbe essere affidabile, accurata e veloce., La precisione della polimerasi o” fedeltà ” si riferisce alla propensione a incorporare il nucleotide corretto come specificato dal filamento del modello. Gli enzimi PCR standard sono, non sorprendentemente, abbastanza precisi. Anche la DNA polimerasi Thermus aquaticus (Taq), considerata una polimerasi PCR a bassa fedeltà, commette un errore solo una volta in circa inserimenti nucleotidici (12)., La scoperta della polimerasi e gli sforzi ingegneristici hanno prodotto polimerasi ad alta fedeltà, che raramente commettono errori di sostituzione della base, richiedendo metodi di sequenziamento del DNA per leggere milioni di basi sintetizzate per rilevare eventuali errori da parte della polimerasi I progressi nella misurazione della fedeltà da parte del sequenziamento a singola molecola hanno identificato la DNA polimerasi ad alta fedeltà Q5® ,
Punti di controllo della fedeltà: la selezione geometrica nel sito attivo e oltre
Le DNA polimerasi garantiscono una replica accurata utilizzando una serie di punti di controllo molecolari, nel sito di incorporazione dei nucleotidi e oltre (1). Durante l’addizione del nucleotide, il nucleotide entrante corretto è posizionato per un allineamento produttivo dei gruppi catalitici, assicurando l’incorporazione efficiente. Questo allineamento per la catalisi è sensibile alle distorsioni di posizione causate da un errato accoppiamento di base Watson-Crick, consentendo lo stallo cinetico a coppie di basi errate o non cognate.,
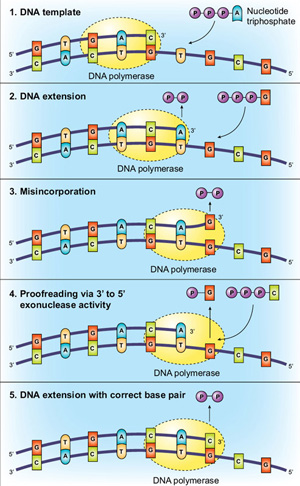
Correzione di bozze di DNA polimerasi
Un altro metodo per aumentare la fedeltà è che la polimerasi abbia 3→5 attività esonucleasica, chiamata “correzione di bozze”. Utilizzando i punti di controllo molecolari base-pairing e sito attivo sopra descritti, Taq DNA polimerasi è incredibilmente preciso, ma enzimi correzione di bozze possono avere ancora più alta fedeltà. Questa precisione aggiuntiva viene trasmessa tramite correzione di bozze, con la polimerasi che “controlla” se il nucleotide corretto è stato inserito nel modello., Se viene rilevata una mancata corrispondenza, il DNA viene trasferito dal dominio di polimerizzazione a un dominio di esonucleasi N-terminale 3→5 della polimerasi. Il nucleotide incorporato in modo errato viene asportato, il DNA torna nel dominio di polimerizzazione e la copia può riprendere (Figura 2).
Il batteriofago T4 si è rivelato un utile sistema sperimentale per valutare l’importanza dell’attività 3→5 dell’esonucleasi per un’accurata replicazione del DNA (6). Sono state identificate mutazioni nel gene T4 43 (che codifica la DNA polimerasi) che hanno diminuito o aumentato la fedeltà. Definendo un rapporto di attività esonucleasi/polimerasi (N/P) per un enzima mutante è stato riscontrato che le polimerasi con bassi rapporti N/P erano più soggette a errori rispetto a quelle con alti rapporti N/P., Una spiegazione per questa osservazione è che al momento dell’incorporazione di una base non corrispondente è più probabile che l’esonucleasi rimuova il nucleotide prima che l’attività della polimerasi la estenda in enzimi con rapporti N/P più elevati. È interessante notare che l’efficacia della correzione di bozze di una polimerasi può mostrare dipendenza dalla sequenza. Ad esempio, le sequenze AT-rich sono corrette in modo più efficace rispetto alle regioni GC. Si ritiene che questa discrepanza sia dovuta alla minore stabilità delle regioni AT che facilita la fusione dei fili e quindi l’attività di correzione delle bozze.,
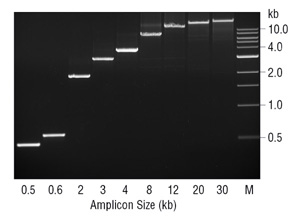
L’assenza di attività esonucleasica 3→5 può avere ramificazioni diverse dalla fedeltà nella PCR. La mancanza di attività di correzione di bozze nella Taq DNA polimerasi è stata proposta per limitare la dimensione dell’amplicone possibile con questo enzima (7). Generalmente, Taq funziona meglio quando amplifica frammenti di DNA < 2 kb e può funzionare con frammenti fino a 3-4 kb. Quando mantenuto a questa dimensione amplicone, Taq è un enzima robusto, facilmente ottimizzato. Tuttavia, sopra ~3 kb scende rapidamente in efficacia., Durante la PCR, Taq disincorporerà i nucleotidi e produrrà disallineamenti, in cui è soggetto a stallo ed è più probabile che si dissocia prima di estendersi rispetto alle 3 estremità accoppiate correttamente. Pertanto, ad una certa dimensione amplicon e polimerasi tasso di errore abbastanza non corrispondenti 3 estremità possono accumularsi per inibire efficacemente il processo PCR. Queste 3 estremità non corrispondenti sono particolarmente problematiche per Taq perché manca l’attività 3→5 esonucleasi per rimuoverle., Aggiungendo una piccola quantità di enzima di correzione di bozze come la DNA polimerasi Deep Vent®, è possibile ottenere un’amplificazione di frammenti ≥ 20 kb (Figura 3). Poiché la stragrande maggioranza dell’enzima nella miscela è la DNA polimerasi Taq, probabilmente sta facendo la maggior parte dell’estensione del primer, con la correzione di bozze della polimerasi Deep Vent che rimuove i disallineamenti inibitori 3 generati da Taq.,
Processività della polimerasi
L’importanza dell’attività di correzione di bozze per la PCR è stata ampiamente conosciuta per quasi due decenni, ma un’altra proprietà, la processività, ha guadagnato maggiore attenzione., “Processività” è un termine che si riferisce al numero di nucleotidi incorporati da una polimerasi in un singolo evento di legame (prima della dissociazione). Taq DNA polimerasi aggiunge circa 50 nucleotidi per evento di legame (8). Perché questo importa? Una polimerasi a bassa processività o “distributiva” estende una popolazione di modelli in modo notevolmente diverso rispetto a una polimerasi processuale. Una polimerasi altamente distributiva si lega a un modello, aggiunge un paio di nucleotidi e si dissocia, lasciando una popolazione di modelli che possono essere estesi ugualmente con il tempo., Una polimerasi altamente processiva lega un modello e si estende con eventi di legame più lunghi.
Ne consegue che, dato abbastanza tempo, il risultato di una reazione di polimerasi processiva o distributiva sarebbe una popolazione di modelli copiati. Tuttavia, in determinate circostanze è possibile che la polimerasi processiva abbia prestazioni superiori. La subunità α della polimerasi III di E. coli, parte della polimerasi replicativa principale, ha una processività di< 10 coppie di basi e una velocità di< 20 nucleotidi/secondo (nt/s)., Tuttavia, quando la subunità si associa alle altre subunità del replisome, in particolare il morsetto scorrevole, la processività effettiva e la velocità di replica aumentano a> 50 kb e 1.000 nt/s, rispettivamente (9). Il termine “processività efficace” è usato perché ci sono dati che indicano che la subunità della polimerasi può scambiare nel replisoma, ma il replisoma mantiene la replicazione del DNA veloce e processuale (10).
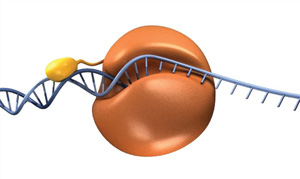
Per sfruttare la processività nella PCR, i ricercatori hanno fuso un dominio di legame del DNA con una polimerasi archaeal (11)., Questo enzima chimerico ha diverse proprietà migliorate, ma in particolare è in grado di amplificare il DNA con tempi di estensione più brevi e produrre prodotti a DNA più lunghi in modo più efficiente, riducendo così i tempi complessivi di termociclaggio. Questa idea di fusione è alla base della DNA polimerasi ad alta fedeltà Q5 e della DNA polimerasi ad alta fedeltà Phusion®, due polimerasi disponibili da NEB (Figura 4).
Direzioni future
Molte proprietà influenzano l’efficacia e l’utilità di una polimerasi PCR. L’architettura del sito attivo della polimerasi e l’attività di correzione delle bozze influenzano l’accuratezza del prodotto finale. Le miscele di polimerasi e la fusione a una proteina legante il DNA conferiscono prestazioni PCR superiori per la lunghezza dell’amplicone e, nel caso della chimera, la velocità di reazione., Altri importanti progressi nella PCR, come le polimerasi hot-start per aumentare la specificità della reazione, la PCR multiplex (Figura 5) e la qPCR hanno anche rivoluzionato le scienze della vita.
Come dimostrato da miscele ingegnerizzate e chimere, le proprietà della polimerasi stessa possono essere modulate per migliorare le prestazioni della PCR. In futuro, è probabile che le proprietà della polimerasi saranno sempre più adattate a specifiche applicazioni PCR e, come tale, questa è un’importante area di ricerca presso NEB.,

Visualizza il grafico di selezione della DNA polimerasi di NEB















