Biology for Majors I (Italiano)
Learning Outcomes
- Descrivi il ruolo della RNA polimerasi
Questa sezione approfondirà il ruolo specifico delle RNA polimerasi durante la trascrizione. Continua a leggere per imparare il ruolo delle RNA polimerasi in ogni fase della trascrizione.,
Inizio della trascrizione
A differenza della polimerasi procariotica che può legarsi a un modello di DNA da solo, gli eucarioti richiedono diverse altre proteine, chiamate fattori di trascrizione, per legarsi prima alla regione del promotore e quindi aiutare a reclutare la polimerasi appropriata.
Le tre RNA polimerasi eucariotiche
Le caratteristiche della sintesi di mRNA eucariotico sono marcatamente più complesse di quelle dei procarioti. Invece di una singola polimerasi comprendente cinque subunità, gli eucarioti hanno tre polimerasi che sono ciascuna composta da 10 subunità o più., Ogni polimerasi eucariotica richiede anche un insieme distinto di fattori di trascrizione per portarlo al modello di DNA.
L’RNA polimerasi I si trova nel nucleolo, una sottostruttura nucleare specializzata in cui l’RNA ribosomiale (rRNA) viene trascritto, elaborato e assemblato in ribosomi (Tabella 1). Le molecole di rRNA sono considerate RNA strutturali perché hanno un ruolo cellulare ma non sono tradotte in proteine. Gli RRNA sono componenti del ribosoma e sono essenziali per il processo di traduzione. L’RNA polimerasi I sintetizza tutti gli RRNA ad eccezione della molecola 5S rRNA., La designazione ” S “si applica alle unità “Svedberg”, un valore non aggiuntivo che caratterizza la velocità con cui una particella si sedimenta durante la centrifugazione.
| Tabella 1.,f Transcription | α-Amanitin Sensitivity | |||
|---|---|---|---|---|
| I | Nucleolus | All rRNAs except 5S rRNA | Insensitive | |
| II | Nucleus | All protein-coding nuclear pre-mRNAs | Extremely sensitive | |
| III | Nucleus | 5S rRNA, tRNAs, and small nuclear RNAs | Moderately sensitive | |
RNA polymerase II is located in the nucleus and synthesizes all protein-coding nuclear pre-mRNAs., I pre-MRNA eucariotici sono sottoposti a un’ampia elaborazione dopo la trascrizione ma prima della traduzione (Figura 1). Per chiarezza, la discussione di questo modulo sulla trascrizione e traduzione negli eucarioti userà il termine “mRNA” per descrivere solo le molecole mature e elaborate che sono pronte per essere tradotte. L’RNA polimerasi II è responsabile della trascrizione della stragrande maggioranza dei geni eucariotici.

Figura 1. L’mRNA eucariotico contiene introni che devono essere impiombati. Un 5′ cap e 3 ‘ poly-A coda sono anche aggiunti.,
L’RNA polimerasi III si trova anche nel nucleo. Questa polimerasi trascrive una varietà di RNA strutturali che include il pre-rRNA 5S, i pre-RNA di trasferimento (pre-tRNA) e i piccoli pre-RNA nucleari. I TRNA hanno un ruolo critico nella traduzione; servono come molecole adattatori tra il modello di mRNA e la catena polipeptidica in crescita. I piccoli RNA nucleari hanno una varietà di funzioni, tra cui “splicing” pre-MRNA e regolazione dei fattori di trascrizione.,
Uno scienziato che caratterizza un nuovo gene può determinare quale polimerasi lo trascrive testando se il gene è espresso in presenza di un particolare veleno di funghi, α-amanitina (Tabella 1). È interessante notare che l’α-amanitina prodotta da Amanita phalloides, il fungo del cappuccio della morte, colpisce le tre polimerasi in modo molto diverso. L’RNA polimerasi I è completamente insensibile all’α-amanitina, il che significa che la polimerasi può trascrivere il DNA in vitro in presenza di questo veleno. Al contrario, l’RNA polimerasi II è estremamente sensibile all’α-amanitina e l’RNA polimerasi III è moderatamente sensibile., Conoscendo la polimerasi trascrizione può indizio un ricercatore nella funzione generale del gene in fase di studio. Poiché l’RNA polimerasi II trascrive la stragrande maggioranza dei geni, ci concentreremo su questa polimerasi nelle nostre discussioni successive sui fattori di trascrizione e sui promotori eucariotici.
Promotori della RNA polimerasi II e fattori di trascrizione
I promotori eucariotici sono molto più grandi e complessi dei promotori procariotici. Tuttavia, entrambi hanno una sequenza simile alla sequenza -10 dei procarioti., Negli eucarioti, questa sequenza è chiamata TATA box e ha la sequenza di consenso TATAAA sul filo di codifica. Si trova a -25 a -35 basi rispetto al sito di iniziazione (+1) (Figura 2). Questa sequenza non è identica alla scatola E. coli -10, ma conserva l’elemento ricco di A–T. La termostabilità dei legami A-T è bassa e questo aiuta il modello di DNA a svolgersi localmente in preparazione per la trascrizione.,
Invece del semplice fattore σ che aiuta a legare l’RNA polimerasi procariotica al suo promotore, gli eucarioti assemblano un complesso di fattori di trascrizione necessari per reclutare l’RNA polimerasi II in un gene codificante la proteina. I fattori di trascrizione che si legano al promotore sono chiamati fattori di trascrizione basali. Questi fattori basali sono tutti chiamati TFII (per il fattore di trascrizione/polimerasi II) più una lettera aggiuntiva (A-J). Il complesso principale è TFIID, che include una proteina legante TATA (TBP)., Gli altri fattori di trascrizione cadono sistematicamente sul modello di DNA, ciascuno stabilizzando ulteriormente il complesso di pre-iniziazione e contribuendo al reclutamento della RNA polimerasi II.
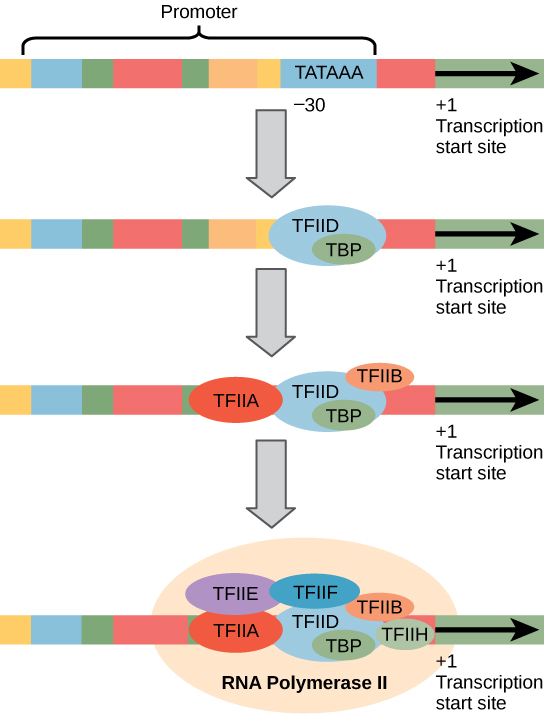
Figura 2. Viene mostrato un promotore generalizzato di un gene trascritto dalla RNA polimerasi II. I fattori di trascrizione riconoscono il promotore. L’RNA polimerasi II poi lega e forma il complesso di inizio della trascrizione.,
Domanda pratica
Uno scienziato giuntura un promotore eucariotico di fronte a un gene batterico e inserisce il gene in un cromosoma batterico. Ti aspetteresti che i batteri trascrivano il gene?
Alcuni promotori eucariotici hanno anche una scatola CAAT conservata (GGCCAATCT) a circa -80. Più a monte della scatola di TATA, i promotori eucariotici possono anche contenere una o più scatole GC-rich (GGCG) o scatole octamer (ATTTGCAT)., Questi elementi legano fattori cellulari che aumentano l’efficienza dell’inizio della trascrizione e sono spesso identificati in geni più “attivi” che vengono costantemente espressi dalla cellula.
I fattori di trascrizione basali sono cruciali nella formazione di un complesso di preiniziazione sul modello del DNA che successivamente recluta la RNA polimerasi II per l’inizio della trascrizione. La complessità della trascrizione eucariotica non termina con le polimerasi e i promotori., Un esercito di altri fattori di trascrizione, che si legano agli esaltatori e ai silenziatori a monte, aiuta anche a regolare la frequenza con cui il pre-mRNA viene sintetizzato da un gene. Esaltatori e silenziatori influenzano l’efficienza della trascrizione, ma non sono necessari per la trascrizione di procedere.
L’evoluzione dei promotori
L’evoluzione dei geni può essere un concetto familiare. Mutazioni possono verificarsi nei geni durante la replicazione del DNA, e il risultato può o non può essere vantaggioso per la cellula., Alterando un enzima, una proteina strutturale o qualche altro fattore, il processo di mutazione può trasformare funzioni o caratteristiche fisiche. Tuttavia, i promotori eucariotici e altre sequenze di regolazione genica possono evolvere pure. Per esempio, si consideri un gene che, nel corso di molte generazioni, diventa più prezioso per la cellula. Forse il gene codifica una proteina strutturale che la cellula ha bisogno di sintetizzare in abbondanza per una certa funzione. Se questo è il caso, sarebbe vantaggioso per la cellula per il promotore di quel gene reclutare i fattori di trascrizione in modo più efficiente e aumentare l’espressione genica.,
Gli scienziati che hanno esaminato l’evoluzione delle sequenze di promotori hanno riportato risultati variabili. In parte, questo è perché è difficile dedurre esattamente dove un promotore eucariotico inizia e finisce. Alcuni promotori si verificano all’interno dei geni; altri si trovano molto a monte, o anche a valle, dei geni che stanno regolando. Tuttavia, quando i ricercatori hanno limitato il loro esame alle sequenze del promotore del centro umano che sono state definite sperimentalmente come sequenze che legano il complesso di preinitiation, hanno trovato che i promotori evolvono ancora più velocemente dei geni proteina-codificanti.,
Non è ancora chiaro come l’evoluzione del promotore possa corrispondere all’evoluzione degli esseri umani o di altri organismi superiori. Tuttavia, l’evoluzione di un promotore per rendere efficacemente più o meno di un dato prodotto genetico è un’alternativa intrigante all’evoluzione dei geni stessi.
Strutture promotrici per RNA polimerasi I e III
I processi di portare RNA polimerasi I e III al modello di DNA coinvolgono raccolte leggermente meno complesse di fattori di trascrizione, ma il tema generale è lo stesso.,
Gli elementi del promotore conservati per i geni trascritti dalle polimerasi I e III differiscono da quelli trascritti dalla RNA polimerasi II. L’RNA polimerasi I trascrive geni che hanno due sequenze di promotori ricche di GC nella regione da -45 a +20. Queste sequenze da sole sono sufficienti per l’inizio della trascrizione, ma i promotori con sequenze aggiuntive nella regione da -180 a -105 a monte del sito di inizio aumenteranno ulteriormente l’inizio. I geni che sono trascritti dalla RNA polimerasi III hanno promotori a monte o promotori che si verificano all’interno dei geni stessi.,
La trascrizione eucariotica è un processo strettamente regolato che richiede una varietà di proteine per interagire tra loro e con il filamento di DNA. Sebbene il processo di trascrizione negli eucarioti comporti un maggiore investimento metabolico rispetto ai procarioti, assicura che la cellula trascriva precisamente i pre-mRNA di cui ha bisogno per la sintesi proteica.,
Allungamento e terminazione
Dopo la formazione del complesso di preiniziazione, la polimerasi viene rilasciata dagli altri fattori di trascrizione e l’allungamento può procedere come nei procarioti con la polimerasi che sintetizza il pre-mRNA nella direzione da 5 ‘a 3’. Come discusso in precedenza, l’RNA polimerasi II trascrive la quota maggiore di geni eucariotici, quindi questa sezione si concentrerà su come questa polimerasi realizza l’allungamento e la terminazione.,
Sebbene il processo enzimatico di allungamento sia essenzialmente lo stesso negli eucarioti e nei procarioti, il modello del DNA è più complesso. Quando le cellule eucariotiche non si dividono, i loro geni esistono come una massa diffusa di DNA e proteine chiamate cromatina. Il DNA è impacchettato strettamente intorno alle proteine cariche dell’istone agli intervalli ripetuti. Questi complessi di DNA-istone, chiamati collettivamente nucleosomi, sono regolarmente distanziati e includono 146 nucleotidi di DNA avvolti intorno a otto istoni come filo attorno a una bobina.,
Affinché si verifichi la sintesi del polinucleotide, il macchinario di trascrizione deve spostare gli istoni fuori strada ogni volta che incontra un nucleosoma. Ciò è compiuta da un complesso speciale della proteina chiamato FATTO, che sta per “facilita la trascrizione della cromatina.”Questo complesso allontana gli istoni dal modello di DNA mentre la polimerasi si muove lungo di esso. Una volta sintetizzato il pre-mRNA, il complesso FACT sostituisce gli istoni per ricreare i nucleosomi.
La terminazione della trascrizione è diversa per le diverse polimerasi., A differenza dei procarioti, l’allungamento da RNA polimerasi II negli eucarioti avviene 1.000-2.000 nucleotidi oltre la fine del gene che viene trascritto. Questa coda pre-mRNA viene successivamente rimossa dalla scissione durante l’elaborazione dell’mRNA. D’altra parte, le RNA polimerasi I e III richiedono segnali di terminazione. I geni trascritti dalla RNA polimerasi I contengono una sequenza specifica di 18 nucleotidi che viene riconosciuta da una proteina di terminazione. Il processo di terminazione nella RNA polimerasi III comporta una forcina mRNA simile alla terminazione indipendente da rho della trascrizione nei procarioti.,
Provalo
Contribuisci!
Migliora questa paginaimpara di più
- H Liang et al., “Fast evolution of core promoters in primate genomes,” Molecular Biology and Evolution 25 (2008): 1239-44. ↵















