Anatomia de uma função e estrutura polimerase – como estão relacionadas
a replicação precisa do genoma é fundamental para a viabilidade de qualquer organismo. O conceito geral para copiar o DNA era evidente sobre a elucidação da estrutura helicoidal dupla do DNA e a identificação da complementaridade do par base (1): uma cadeia de nucleobases poderia servir como modelo para a síntese de uma nova cadeia. Dentro de uma década dessas descobertas, um agente foi purificado a partir de E. coli que catalisou a duplicação da cadeia de DNA (2). Este agente foi chamado de “polimerase”. E., coli DNA polimerase I, a primeira polimerase de DNA descoberta não foi a polimerase replicativa primária, mas sim uma envolvida na resolução de fragmentos de strand Okazaki e reparação de DNA. Isso prenunciou futuras descobertas de muitas famílias de DNA polimerase, cada uma servindo requisitos celulares específicos para a replicação e reparação do DNA.as polimerases do ADN servem como enzimas fundamentais nas ciências da vida pela mesma razão que são de natureza crítica: copiam o ADN. Aplicações de polimerase incluem rotulagem de DNA, sequenciação e amplificação., Um protocolo específico de amplificação, a reação em cadeia da polimerase (PCR) é uma técnica amplamente utilizada que emprega polimerases termofílicas para amplificar exponencialmente segmentos específicos de DNA (3) e permite uma gama de aplicações desde diagnósticos humanos e patógenos à clonagem molecular em laboratórios de biologia ao redor do mundo.
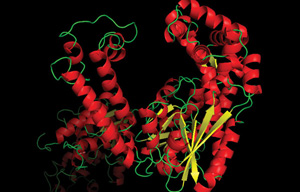
Polimerase Precisão
PCR põe as mesmas demandas básicas em um polimerase como uma célula coloca no seu sistema de replicação. Essencialmente, a polimerase deve ser confiável, precisa e rápida., Precisão da polimerase ou” fidelidade ” refere-se à propensão para incorporar o nucleótido correto, conforme especificado pela cadeia de template. As enzimas PCR padrão são, não surpreendentemente, bastante precisas. Mesmo a TerMus aquaticus (Taq) ADN polimerase, considerada uma PCR polimerase de baixa fidelidade, só comete um erro uma vez em inserções de nucleótidos (12)., A descoberta e os esforços de engenharia da polimerase produziram polimerases de alta fidelidade, que raramente cometem erros de substituição de base, exigindo métodos de sequenciação de DNA para ler milhões de bases sintetizadas para detectar quaisquer erros dos avanços da polimerase na medição da fidelidade pela sequenciação de uma única molécula identificou a polimerase de alta fidelidade Q5® como tendo uma fidelidade 280X maior da polimerase de thanTaq (12).,testes de fidelidade: a selecção geométrica no local activo e para além das polimerases do ADN assegura a replicação exacta utilizando uma série de testes moleculares, no local de incorporação dos nucleótidos e para além (1). Durante a adição de nucleótidos, o nucleótido de entrada correto é posicionado para um alinhamento produtivo de grupos catalíticos, garantindo uma incorporação eficiente. Este alinhamento para catálise é sensível a distorções na posição causadas por pares de base Watson-Crick incorretos, permitindo emparelhamento cinético em pares de base incorretos ou não cognatos.,
Proofreading of DNA Polymerases
Another method of increasing fidelity is for the polymerase to have 3→5 exonuclease activity, named “proofreading”. Usando os pontos de verificação moleculares de base e local ativo descritos acima, a Taq DNA polimerase é incrivelmente precisa, mas as enzimas de revisão podem ter uma fidelidade ainda maior. Esta precisão adicional é transmitida através de revisão, com a polimerase “verificando” se o nucleótido correto foi inserido no modelo., If a mismatch is detected the DNA is transferred from the polymerization domain to an n-terminal 3→5 exonuclease domain of the polymerase. O nucleótido incorrectamente incorporado é excisado, o ADN volta para o domínio da polimerização e a cópia pode ser retomada (Figura 2).
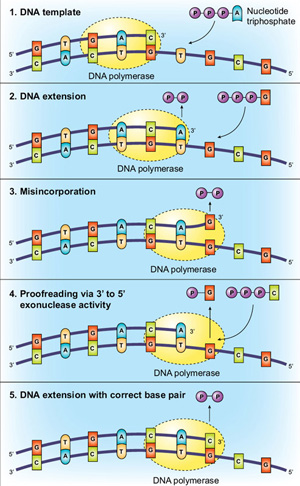
Bacteriophage T4 proved to be a useful experimental system for evaluating the importance of 3→5 exonuclease activity for accurate DNA replication (6). Foram identificadas mutações no gene T4 43 (que codifica a ADN polimerase) que diminuíram ou aumentaram a fidelidade. Ao definir uma razão de actividade da exonuclease/polimerase (n/P) para uma enzima mutante, verificou-se que as polimerases com rácios n/P baixos eram mais propensas a erros do que aquelas com rácios n / P elevados., Uma explicação para esta observação é que após a incorporação de uma base inigualável, é mais provável que a exonuclease remova o nucleótido antes que a atividade da polimerase a estenda em enzimas com razões N/P mais elevadas. Curiosamente, a eficácia de revisão de uma polimerase pode mostrar dependência de sequência. Por exemplo, sequências ricas em AT são mais efetivamente corrigidas do que as regiões GC. Pensa-se que esta discrepância se deve à menor estabilidade das regiões de TA, que facilita a fusão da vertente e, por conseguinte, a actividade de revisão.,
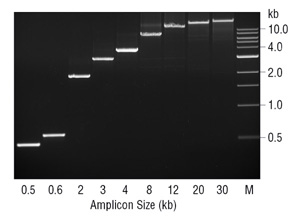
a ausência de 3→5 atividade de exonuclease pode ter ramificações diferentes da fidelidade na PCR. A falta de actividade de revisão da ADN polimerase Taq foi proposta para limitar o tamanho do fragmento amplificado possível com esta enzima (7). Geralmente, Taq tem melhor desempenho ao amplificar fragmentos de DNA < 2 kb, e pode trabalhar com fragmentos até 3-4 kb. Quando mantido a este tamanho de amplidão, Taq é uma enzima robusta e facilmente otimizada. No entanto, acima de ~3 kb ele cai rapidamente em eficácia., Durante a PCR, Taq irá misincorporar nucleótidos e produzir mismatches, em que é propenso a empatar e é mais provável a dissociar antes de se estender em comparação com corretamente base emparelhada 3 extremidades. Portanto, a um certo tamanho de amplicão e taxa de erro de polimerase bastante incompatíveis 3 extremidades podem se acumular para efetivamente inibir o processo de PCR. Estes 3 extremos inadequados são particularmente problemáticos para Taq porque não tem a atividade de 3→5 exonucleases para removê-los., Adicionando uma pequena quantidade de enzima de proofreading, como a DNA polimerase profunda Vent®, pode-se obter a amplificação de fragmentos ≥ 20 kb (Figura 3). Uma vez que a grande maioria da enzima na mistura é a Taq ADN polimerase, provavelmente está a fazer a maior parte da extensão da primer, com a proofreading polimerase profunda do ventilador a remover os 3 desmatamentos inibitórios gerados pela Taq.,
Polimerase Processivity
A importância da atividade de revisão para a PCR tem sido amplamente conhecido por quase duas décadas, mas outro imóvel, processivity, ganhou maior atenção., “Processividade” é um termo que se refere ao número de nucleótidos incorporados por uma polimerase em um único evento de ligação (antes da dissociação). A TAQ ADN polimerase adiciona aproximadamente 50 nucleótidos por evento de ligação (8). Porque é que isso importa? Uma polimerase de baixa processividade ou “distributiva” estende uma população de modelos de uma maneira visivelmente diferente de uma polimerase processiva. Uma polimerase altamente distributiva liga-se a um modelo, adiciona um par de nucleótidos, e dissocia, deixando uma população de modelos que podem ser estendidos igualmente com o tempo., Uma polimerase altamente processiva liga-se a um modelo e estende-se com eventos de ligação mais longos.
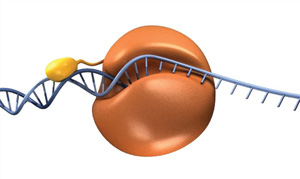
segue-se que, dado o tempo suficiente, o resultado de uma reação de polimerase processiva ou distributiva seria uma população de modelos copiados. No entanto, em certas circunstâncias, é possível que a polimerase processiva tenha um desempenho superior. A E. coli polimerase III subunidade α, parte do principal replicação polimerase, tem um processivity de < 10 pares de base e uma velocidade de < 20 nucleotídeos/segundo (nt/s)., No entanto, quando a subunidade associa-se com as outras subunidades replisome, particularmente o grampo deslizante, a processividade efetiva e velocidade de replicação aumenta para > 50 kb e 1.000 nt/s, respectivamente (9). O termo “processividade efetiva” é usado porque existem dados que indicam que a subunidade da polimerase pode trocar no replisome, mas o replisome mantém a replicação de DNA processiva rápida (10).
para tirar partido da processividade na PCR, os investigadores fundiram um domínio de ligação do ADN a uma polimerase archaeal (11)., Esta enzima quimérica tem várias propriedades melhoradas, mas notavelmente é capaz de amplificar o DNA com tempos de extensão mais curtos e produzir produtos de DNA mais longos de forma mais eficiente, reduzindo assim os tempos de termociclagem geral. Esta ideia de fusão é a base da polimerase de ADN de Alta Fidelidade Q5 e da polimerase de ADN de Alta Fidelidade Phusion®, duas polimerases disponíveis na NEB (Figura 4).
Future Directions
muitas propriedades afetam a eficácia e utilidade de uma polimerase PCR. A arquitectura activa do local da polimerase e a actividade de revisão afectam a exactidão do produto final. A polimerase mistura e fusão a uma proteína de ligação ao ADN confere um desempenho PCR superior para o comprimento do fragmento amplificado e, no caso da Quimera, a velocidade de reacção., Outros avanços importantes na PCR, tais como polimerases de arranque a quente para aumentar a especificidade da reação, PCR multiplex (Figura 5) e qPCR também revolucionaram as ciências da vida. como demonstrado por misturas de engenharia e quimeras, as propriedades da polimerase em si podem ser moduladas para melhorar o desempenho da PCR. No futuro, é provável que as propriedades da polimerase sejam cada vez mais adaptadas a aplicações específicas da PCR e, como tal, esta é uma área importante de pesquisa na NEB.,

View NEB’s DNA Polymerase Selection Chart















